在过去的十年里,深度学习在一系列的应用中取得了巨大的成功。然而,为了验证和可解释性,我们不仅需要模型做出的预测,还需要知道它在做出预测时的置信度。这对于让医学影像学的临床医生接受它是非常重要的。在这篇博客中,我们展示了我们在韦洛尔理工学院进行的研究。我们使用了一个基于变分推理技术的编码解码架构来分割脑肿瘤图像。我们比较了U-Net、V-Net和FCN等不同的主干架构作为编码器的条件分布采样数据。我们使用Dice相似系数(DSC)和IOU作为评价指标来评价我们在公开数据集BRATS上的工作。
医学图像分割
在目前的文献中主要利用两种技术成功地解决了医学图像的分割问题,一种是利用全卷积网络(FCN),另一种是基于U-Net的技术。FCN体系结构的主要特点是在最后没有使用已成功用于图像分类问题的全连接层。另一方面,U-Net使用一种编码器-解码器架构,在编码器中有池化层,在解码器中有上采样层。
贝叶斯神经网络
这是一种可扩展的避免神经网络过拟合的方法,同时也给了我们一个不确定性的度量。神经网络学习给定的数据集的后验分布的权重,而不是基于点的估计,如下面的公式所示。
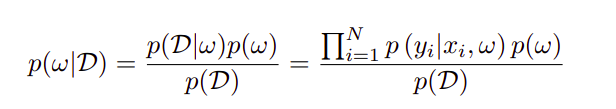
预测分布可以通过逼近积分来计算,如下式所示。
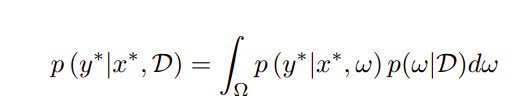
变分推断
变分推断通过最大化证据下界来寻找分布的参数。ELBO由前后分布的Kullback-Leibler (KL)散度和负对数似然(NLL)两项之和构成。需要最小化的KL散度项如下式所示。
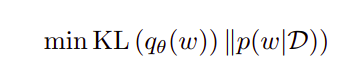
KL散度定义如下式。
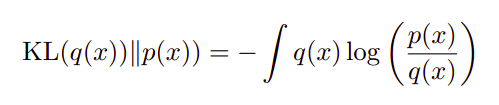
由于上述方程中的积分在本质上是难以处理的,它可以写成另一种形式。该方程可转化为优化问题,如下式所示。
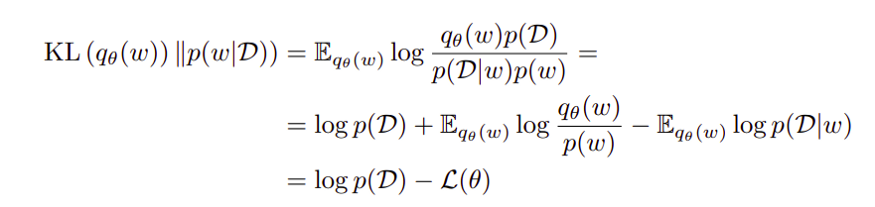
随机不确定性和认知不确定性
有两种类型的不确定性 —— 随机不确定性和认知不确定性,其中方差是两者的总和。对于最终的预测,单个的均值和方差可以估计,如下两个方程所示。
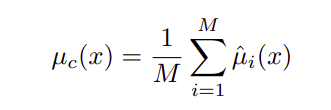
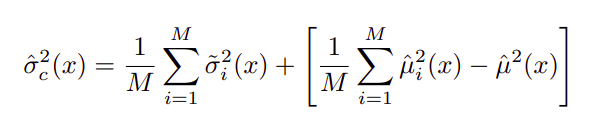
方差中的第一项表示随机不确定性,而第二项表示认知不确定性。
网络结构
先验分布有助于整合网络上的权值学习。我们的模型使用了与VAEs中使用的类似的编码器解码器体系结构,编码器的输入来自预先训练好的图像分割结构。输入到编码器只需要表示置信度的条件分布的标准差向量的均值,以此来正确预测像素点。参数经过编码器后,被转换为一个潜在表示,再采样的平均值和标准偏差向量。解码器随后将其恢复到原始分布。采用传统的反向传播算法进行梯度下降模型的训练。本工作中使用的模型架构如图1所示:
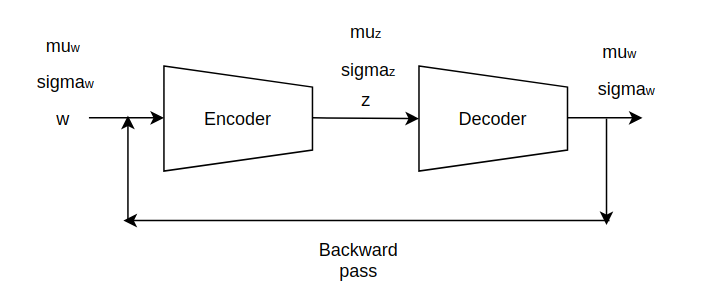
图1:模型结构
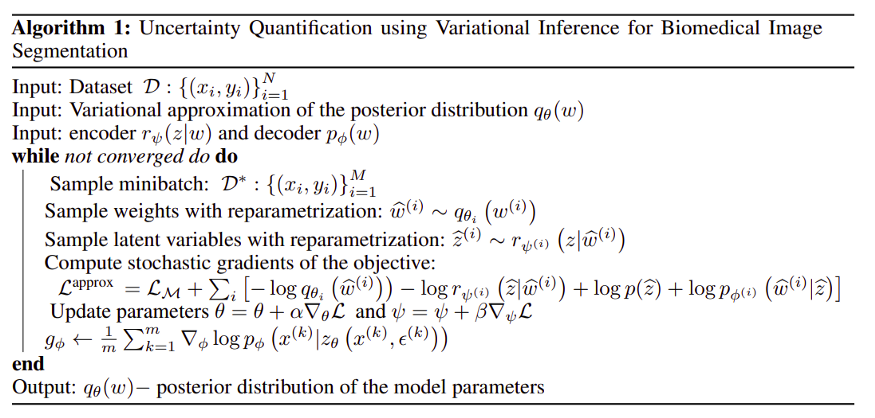
算法
下面是基于随机梯度下降的训练网络的算法。
数据集
为了评估我们的网络性能,我们使用BRATS18脑瘤分割数据集。它包含175名恶性胶质瘤和低级别恶性胶质瘤患者的MRI扫描。图像分辨率为240×240×155像素。ground truth标签是由神经放射学专家创建的。数据集的一个示例如图2所示。
图2:MRI切片的例子以及分割的ground truth
评估指标
评价指标为Dice相似系数(DSC),也称F1-score和IoU。对应的方程如下所示。
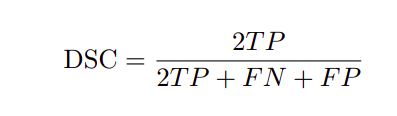
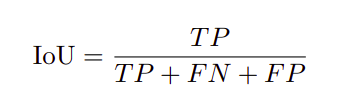
损失函数
采用二元交叉熵和dice损失相结合的方法对网络进行训练。第一部分二元交叉熵是分类问题中常用的损失函数,如下式所示:
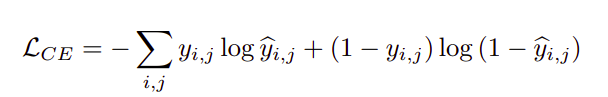
二元交叉熵损失的问题在于它没有考虑到类的不平衡,因为背景是占主导地位的类。dice损失解决了这个问题,可以写成如下公式。
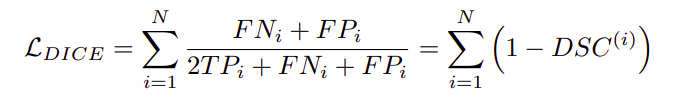
这两个损失项被合并在一个项中,并给予dice损失项更多的权重,因为它能更好地处理类别不平衡问题。这是用下面的公式定义的。
结果
分割所涉及的不确定性如图3所示。深的颜色表示更自信,而浅的颜色表示模型在这些区域不太自信。
图3:与ground truth分割相比,测试样本上的模型预测示例。第一列:输入图像,第二列:真值分割,第三列:预测分割,第四列:随机不确定性,第五列:认知不确定性
总结
在这个博客中,我们提出了一种在医学图像分割中量化不确定性的方法。我们的模型基于一个类似于VAEs所使用的编码器解码器框架。网络的权值代表分布而不是点估计,从而在进行预测的同时给出了一种原则性的测量不确定性的方法。编码器的输入来自于预训练的骨干架构,如U-Net, V-Net, FCN,这些架构都是从条件分布中采样的,代表了像素被正确标记的置信度。我们在公开数据集BRATS上评估我们的结果,使用DSC和IOU指标,我们的网络优于以前的SOTA的结果。
原文标题:解析丨对医学图像分割中的置信度进行量化
文章出处:【微信公众号:通信信号处理研究所】欢迎添加关注!文章转载请注明出处。
责任编辑:haq
-
神经网络
+关注
关注
42文章
4774浏览量
100912 -
深度学习
+关注
关注
73文章
5507浏览量
121299
原文标题:解析丨对医学图像分割中的置信度进行量化
文章出处:【微信号:tyutcsplab,微信公众号:智能感知与物联网技术研究所】欢迎添加关注!文章转载请注明出处。
发布评论请先 登录
相关推荐
语义分割25种损失函数综述和展望

图像语义分割的实用性是什么
图像分割与目标检测的区别是什么
机器学习中的数据分割方法
图像分割与语义分割中的CNN模型综述
机器人视觉技术中图像分割方法有哪些
常见的医学图像读取方式和预处理方法
基于门控线性网络(GLN)的高压缩比无损医学图像压缩算法
Harvard FairSeg:第一个用于医学分割的公平性数据集





 如何对医学图像分割中的置信度进行量化?
如何对医学图像分割中的置信度进行量化?











评论