使用 NVIDIA Clara 构建的医疗成像 AI 模型现在可以在云计算的 MD.ai 上本地运行,从而使用现代 web 浏览器实现协作模型验证和快速注释项目。这些 NVIDIA Clara 模型可免费用于任何 MD.ai 合作研究项目,如器官或肿瘤分割。
人工智能解决方案已被证明有助于简化放射学和企业成像工作流程。然而,创建、共享、测试和缩放计算机视觉模型的过程并不像所有模式、条件和结果那样简化。需要几个关键组件来创建稳健的模型,并支持最多样化的采集设备和患者群体。这些关键组件可以包括为未注成像研究创建基本事实的能力,以及在全球范围内合作评估模型与验证数据的使用的能力。
MD.ai 的实时协作标注平台和英伟达 Clara 深度学习培训框架有助于创建更健壮的模型构建和协作。
在这篇文章中,我们将介绍 Clara Train MMAR 的基础知识,以及准备使用 MD.ai 所需的步骤。只需几个步骤,您就可以在 MD.ai 上部署这些预训练模型中的任何一个,以实现无缝的基于 web 的评估和协作。在 MD.ai 上部署之后,这些模型可以用于任何现有或新的 MD.ai 项目。
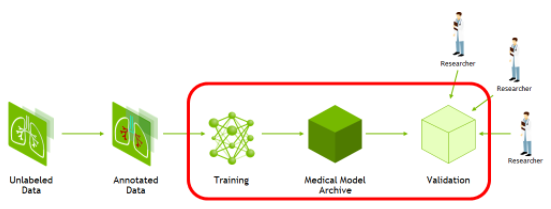
图 1 。培训和验证 AI 模型所需的工作流程 。
NVIDIA Clara Train
Clara 训练框架是一个基于 Python 的应用程序包 NVIDIA Clara Train SDK. 该框架旨在基于 NVIDIA 研究人员内部构建的优化、即用、预训练的医学成像模型,在医学成像领域快速实施深度学习解决方案。
Clara 培训框架使用模型的标准结构 医学模型档案 ( MMAR ),其中包含预培训模型以及定义用于培训、微调、验证和推理的端到端开发工作流的脚本。
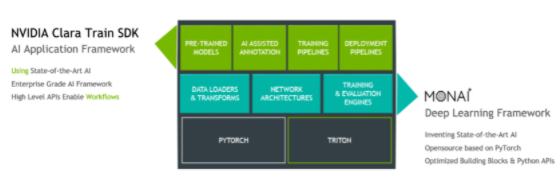
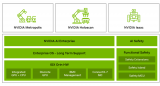
图 2.Clara Train SDK 和 MONAI 深度学习框架高级架构。
Clara Train v4.0 + SDK 使用基于组件的体系结构,该体系结构构建于基于 PyTorch 的开源框架 MONAI (人工智能医疗开放网络) 。 MONAI 提供了医疗成像领域优化的基础功能,可用于在本机 PyTorch 范式中构建培训工作流。 Clara Train SDK 使用这些基础组件(如优化的数据加载程序、转换、丢失函数、优化器和度量)来实现打包为 MMAR 的端到端培训工作流。
MD.ai
MD.ai 提供了一个基于 web 的云本地注释平台,支持临床医生和研究人员团队之间通过共享工作空间进行实时协作。您还可以加载多个深度学习模型以进行实时评估。
该平台为数据集构建和 AI 项目创建提供了一个简单无缝的界面。它为用户提供了一套广泛的工具,用于注释数据和构建机器学习算法,以加速人工智能在医学中的应用,特别关注医学成像。
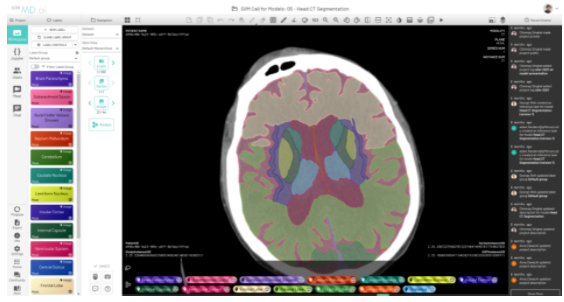
图 3 。显示大脑分割的 MD.ai 用户界面 。
将此功能与在 MD.ai 平台上快速部署 Clara 训练模型 MMAR 的能力相结合,可以为您提供一个端到端的工作流,该工作流涵盖快速模型开发、模型训练、微调、推理以及快速评估和可视化。这种端到端的能力简化了模型从研发到生产的过程。
解决方案概述
Clara Train 的起点是NGC Clara Train 采集。 在这里,您可以找到 Clara Train SDK 容器、一组免费提供的、经过预训练的模型,以及一组 Jupyter 笔记本电脑,其中介绍了 SDK 的主要概念。所有 Clara Train 模型共享前面提到的 MMAR 格式。
Clara Train MMAR 定义了一个标准结构,用于存储定义模型开发工作流所需的文件,以及执行模型进行验证和推断时生成的文件。该结构定义如下:
ROOT config config_train.json config_finetune.json config_inference.json config_validation.json config_validation_ckpt.json environment.json commands set_env.sh train.sh train with single GPU train_multi_gpu.sh train with 2 GPUs finetune.sh transfer learning with CKPT infer.sh inference with TS model validate.sh validate with TS model validate_ckpt.sh validate with CKPT validate_multi_gpu.sh validate with TS model on 2 GPUs validate_multi_gpu_ckpt.sh validate with CKPT on 2 GPUs export.sh export CKPT to TS model resources log.config ... docs license.txt Readme.md ... models model.pt model.ts final_model.pt eval all evaluation outputs: segmentation / classification results metrics reports, etc.
所有提供用于 Clara Train 的预训练模型,以及使用 Clara Train 框架开发的自定义模型都使用此结构。为了准备与 MD.ai 一起使用的 MMAR ,我们假设一个预先训练的模型,并将重点放在部署的几个关键组件上。
第一个组件是 environment.json 文件,它定义了模型的公共参数,包括数据集路径和模型检查点。例如, Clara 序列分段任务中的 environment.json 文件定义了以下参数:
{ "DATA_ROOT": "/workspace/data/Task09_Spleen_nii", "DATASET_JSON": "/workspace/data/Task09_Spleen_nii/dataset_0.json", "PROCESSING_TASK": "segmentation", "MMAR_EVAL_OUTPUT_PATH": "eval", "MMAR_CKPT_DIR": "models", "MMAR_CKPT": "models/model.pt" "MMAR_TORCHSCRIPT": "models/model.ts"
}
准备与 MD.ai 集成的模型时,请确保 MMAR 的models/目录中包含经过培训的MMAR_CKPT和MMAR_TORCHSCRIPT。它们分别通过执行捆绑的train.sh和export.sh生成。
-
train.sh脚本执行模型训练,这需要输入数据集使用DATA_ROOT和DATASET_JSON,并生成 MMAR _ CKPT 。 -
infer.sh脚本将此检查点序列化到用于推断的MMAR_TORCHSCRIPT中。
对于预训练模型,提供了检查点和 TorchScript ,您可以关注推理管道。使用 MMAR 的infer.sh脚本执行推断:
1 #!/usr/bin/env bash 2 my_dir="$(dirname "$0")" 3 . $my_dir/set_env.sh 4 echo "MMAR_ROOT set to $MMAR_ROOT" 5 6 CONFIG_FILE=config/config_validation.json 7 ENVIRONMENT_FILE=config/environment.json 8 python3 -u -m medl.apps.evaluate \ 9 -m $MMAR_ROOT \ 10 -c $CONFIG_FILE \ 11 -e $ENVIRONMENT_FILE \ 12 --set \ 13 DATASET_JSON=$MMAR_ROOT/config/dataset_0.json \ 14 output_infer_result=true \ 15 do_validation=false
此脚本对environment.json中定义的完整数据集的config_validation.json中定义的验证子集进行推断。如果参考测试数据随 MMAR 一起提供,则必须定义该数据的路径。集成 MMAR 时, MD.ai 直接处理数据集,这些值作为集成的一部分被覆盖。
要在 MD.AI 上部署您自己的预训练 AI 模型进行推理,您必须已经有一个现有项目或在平台上创建一个新项目。项目还必须包含用于测试模型的数据集。有关更多信息,请参阅 立项 。
接下来,要部署 AI 模型,必须将推理代码转换为与平台兼容的特定格式。以下文件是成功部署的最低要求:
config.yaml
mdai_deploy.py
requirements.txt
model-weights
对于 NVIDIA Clara 型号,我们已为您进一步简化了此功能,无需从头开始编写这些文件。我们为 NGC 目录支持的每个不同类别的深度学习模型提供了框架代码:分类、分段等。您可以下载特定于模型的框架代码,进行一些调整,这些调整将在本文后面介绍,然后将模型上传到 MD.ai 上进行推断。
推理步骤
准备好 MMAR 后,下面介绍如何直接使用它在 MD.ai 上运行模型。这篇文章将引导您了解一个已经部署在平台上的示例细分模型 在 MD.ai 上运行分段模型的框架代码 ,它实际上是 NVIDIA 的 CT spleen segmentation 模型的代码。
现在,要使用相同的 MMAR 格式部署 肝脏和肿瘤分割 模型,请执行以下步骤:
- 下载分割模型的框架代码。
- 从 NGC 目录下载用于肝脏和肿瘤分割模型的 MMAR。
-
在下载的框架代码中,用下载的 MMAR 文件夹替换
/workspace/clara_pt_spleen_ct_segmentation_1文件夹。 -
在
/workspace/config_mdai.json文件中,进行以下更改:
1 {
2 “type” : “segmentation”,
3 “root_folder”: “clara_pt_spleen_ct_segmentation_1”,
4 “out_classes” : 2,
5 “data_list_key”: “test”
6 }
- root_folder– 将此键值替换为下载的 MMAR 文件夹的名称,例如肝脏和肿瘤示例的 clara _ pt _ liver _和_ tumor _ ct _ segmentation _ 1 。
- out_classes– 将此值替换为模型的输出类数,如本例中的 3 (背景: 0 、肝脏: 1 和肿瘤: 2 )。
- data_list_key– 替换为 MMAR 的 config / config _ interference.json 文件的 data _ list _ key 属性中提到的密钥名称,例如 testing 。
-
在
/mdai文件夹中,进行以下更改:
-
在
config.yaml文件,中,将clara_version键更改为您的模型使用的适当版本(例如, 3.1.01 或 4.0 )。
1 base_image: nvidia 2 clara_version: 4.0 3 device_type: gpu
- 在[EZX29 ],中添加了任何额外的依赖项,比英伟达 Clara 基本图像和文件中已经存在的那些依赖性要多。
这将为您的模型在 MD.ai 上的部署做好准备。脾脏和肝脏肿瘤分割模型都已部署在 MD.ai 平台上,并且是 可供评估 。
分类模型也可以执行类似的步骤,尽管我们正在努力进一步简化这种集成。有关将胸部 X 光分为 15 种异常的示例 NVIDIA 模型的骨架代码,请参见 GitHub 上的 示例:用于胸部 x 光疾病分类的 NVIDIA MMAR 。该型号也是 部署在公共站点上 。
当代码准备就绪时,必须将其包装在 zip 文件中,以便将其上载到 MD.ai 上进行推断。
您的模型现在可以在 MD.ai 中对您选择的任何数据集进行尝试了!
最好的是,所有模型只需在MD.ai平台上部署一次。一旦NVIDIA Clara 模型部署到MD.ai上,它就可以通过使用模型克隆功能在任何MD.ai项目中使用。下面是一个例子,通过复制[EZX37 ]值,将英伟达肝分割模型克隆为一个新的MD AI项目:
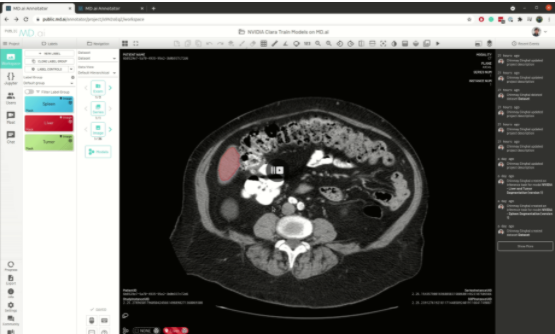 图 5 。基于 MD.ai 的模型克隆工作流.
图 5 。基于 MD.ai 的模型克隆工作流.未来特征
我们正致力于简化集成,以尽量减少部署 MMAR 所需的步骤,并计划消除所有代码修改,以便只需单击一个按钮即可轻松部署。
MD.ai 计划预先部署 NGC 上可用的所有模型,以便您可以通过从我们的公共项目克隆直接使用它们,从而避免您自己部署 MMAR 的过程。我们还将创建 NVIDIA Clara 入门包,这样您就可以轻松地开始使用预装到项目中的选定型号。
另一个重要的计划是增加对在 MD.AI 上训练 AI 模型的支持。当我们有了这个平台,您可以有效地使用英伟达 AI 辅助注释产品在平台上帮助用户注释得更快更容易,而不是从头开始。
总结
在本文中,我们重点介绍了每个平台的关键组件以及在 MD.ai 上快速部署使用 NVIDIA Clara 构建的医疗成像模型所需的步骤。
关于作者
Chinmay Singhal 是 MD.ai 的机器学习工程师。他的工作围绕 MD.ai 机器学习模型部署平台的设计和开发展开。他在纽约大学获得了数据科学硕士学位,专注于深度学习。
Leon Chen 是 MD.ai 的联合创始人兼首席执行官,负责构建医疗 ai 的未来 IDE 。他是哈佛大学培训的内科医生、 Kaggle 硕士、 Keras.js 等流行开源软件的创造者和电子音乐家。
Kris Kersten 是 NVIDIA 的解决方案架构师,专注于 AI ,致力于扩展 ML 和 DL 解决方案,以解决当今医疗领域最紧迫的问题。在加入 NVIDIA 之前, Kris 曾在 Cray 超级计算机公司工作,研究从低级缓存基准测试到大规模并行模拟的硬件和软件性能特征。
审核编辑:郭婷
-
NVIDIA
+关注
关注
14文章
5025浏览量
103268 -
云计算
+关注
关注
39文章
7840浏览量
137561 -
AI
+关注
关注
87文章
31155浏览量
269496
发布评论请先 登录
相关推荐
NVIDIA推出面向RTX AI PC的AI基础模型
NVIDIA推出多个生成式AI模型和蓝图
NVIDIA推出全新生成式AI模型Fugatto
使用NVIDIA AI平台确保医疗数据安全
NVIDIA AI助力实现更好的癌症检测
NVIDIA RTX AI套件简化AI驱动的应用开发
NVIDIA AI Foundry 为全球企业打造自定义 Llama 3.1 生成式 AI 模型

揭秘NVIDIA AI Workbench 如何助力应用开发
NVIDIA 通过 Holoscan 为 NVIDIA IGX 提供企业软件支持,实现边缘实时医疗、工业和科学 AI 应用





 NVIDIA Clara医疗成像AI模型在MD.ai项目中的应用
NVIDIA Clara医疗成像AI模型在MD.ai项目中的应用










评论