上周四,何博士为大家在北鲲云的直播间分享了Amber热力学积分计算相对自由能变化)。
直播结束后有很多小伙伴来向我们要PPT资料,这里何博士也为大家准备了文字版本的教程。将为大家介绍如何在北鲲云计算平台上利用Amber热力学积分计算相对自由能变化,体系包括小分子-蛋白(小分子改变),小分子-蛋白(蛋白突变),蛋白-蛋白相互作用。
本教程要求使用者一定程度了解Amber动力学模拟程序。
Amber是美国加州大学Peter Kollman等开发的一款著名的分子动力学模拟软件包。Amber主要适用于蛋白质,小分子和多糖等生物分子体系的模拟。
本文所需的所有文件请在https://github.com/Xinheng-He/ti_toturial上下载。

该应用场景解决将蛋白口袋内的小分子A变为小分子B所产生的相对自由能变。
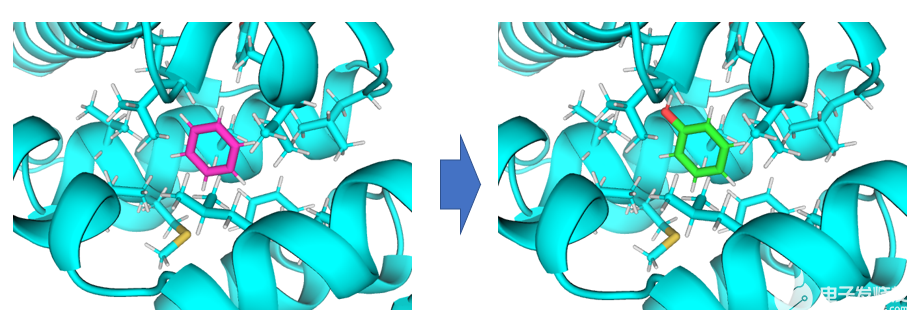
将蛋白口袋内的苯转化为苯酚。
首先,使用pymol将分子打开,并选中小分子,保存为mol2文件,如下图所示,我们使用
save*my_caseben_ligand.mol2save*my_casebenfen_ligand.mol2
这两个命令保存了变化前后的配体。
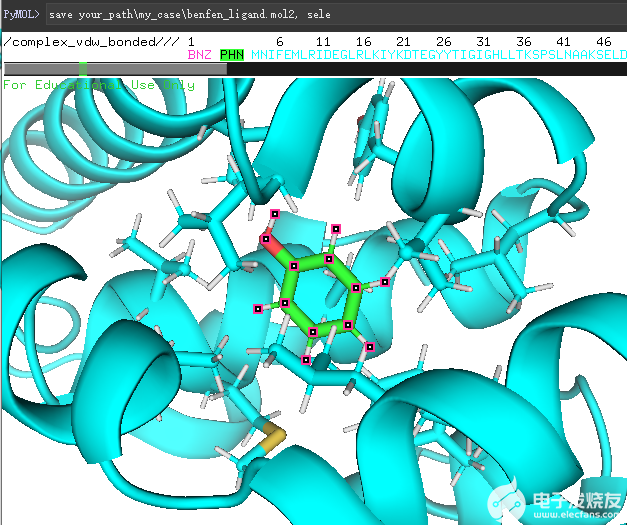
(保存pymol中的sele对象)
开启一个北鲲云管理节点加载环境。
moduleaddAnaconda3/2020.02 source/public/software/.local/easybuild/software/amber/aber20/amber.sh ulimit-sunlimited ulimit-lunlimited
对苯生成具有电荷的可用mol2文件,总电荷为0,残基名为BEN。
antechamber-iben_ligand.mol2-fimol2-oben_real.gaff2.mol2-fomol2-rnBEN-atgaff2-anyes-drno-pfyes-cbcc-nc0
生成frcmod力场参数文件。
parmchk2-iben_real.gaff2.mol2-fmol2-oBEN.gaff2.frcmod-sgaff2-ayes
上述操作对苯酚再来一次。
antechamber-ibenfen_ligand.mol2-fimol2-obenfen_real.gaff2.mol2-fomol2-rnFEN-atgaff2-anyes-drno-pfyes-cbcc-nc0 parmchk2-ibenfen_real.gaff2.mol2-fmol2-oFEN.gaff2.frcmod-sgaff2-ayes
根据frcmod文件,生成两个分子的文库文件,该文件描述了分子内部的原子类型和键连信息。
tleap-f-<<_EOF source leaprc.gaff2 loadamberparams FEN.gaff2.frcmod FEN = loadmol2 benfen_real.gaff2.mol2 saveoff FEN FEN.lib savepdb FEN FEN.pdb quit _EOF tleap -f - <<_EOF source leaprc.gaff2 loadamberparams BEN.gaff2.frcmod BEN = loadmol2 ben_real.gaff2.mol2 saveoff BEN BEN.lib savepdb BEN BEN.pdb quit _EOF
注意前后的力场要保持一致。
将两个pdb文件(FEN.pdb和BEN.pdb)中同样位置的全部重原子,保存成同样的坐标,注意名字要和lib中的一样,放成一个lig.pdb,在下面的tleap过程中,tleap会自动根据lib文件将complex中的原子变成真实的样子,这样做是为了保证一样原子的位置完全一致,减少不必要的变量。
(pdb文件的内容)
使用pdb4amber,检查蛋白是否有二硫键,或需要编辑的残基。
pdb4amber pure_protein.pdb -o pure_check.pdb cat pure_check_sslink
没有二硫键,之后使用pure_check.pdb。
接下来在tleap中加载配体和受体。
tleap-f-<<_EOF
source leaprc.protein.ff14SB
source leaprc.gaff2
source leaprc.water.tip3p
loadAmberParams frcmod.ionsjc_tip3p
loadoff BEN.lib
loadoff FEN.lib
loadamberparams BEN.gaff2.frcmod
loadamberparams FEN.gaff2.frcmod
ligands = loadpdb lig.pdb
complex = loadpdb pure_check.pdb
complex = combine {ligands complex}
check complex
solvatebox ligands TIP3PBOX 15
addions ligands Na+ 0
savepdb ligands ligands_vdw_bonded.pdb
saveamberparm ligands ligands_vdw_bonded.parm7 ligands_vdw_bonded.rst7
solvatebox complex TIP3PBOX 15
addions complex Cl- 0
savepdb complex complex_vdw_bonded.pdb
saveamberparm complex complex_vdw_bonded.parm7 complex_vdw_bonded.rst7
quit
_EOF
注意根据实际电荷情况调整addions,如果ligand/complex带负电,加Na+,反之加Cl-,离子类型可以自己选择。
下载complex_vdw_bonded.pdb并检查其中的配体分子区别,是否只是不一样的配体部分不一样,确定配体不一样部分的原子。设置出发和结束原子。
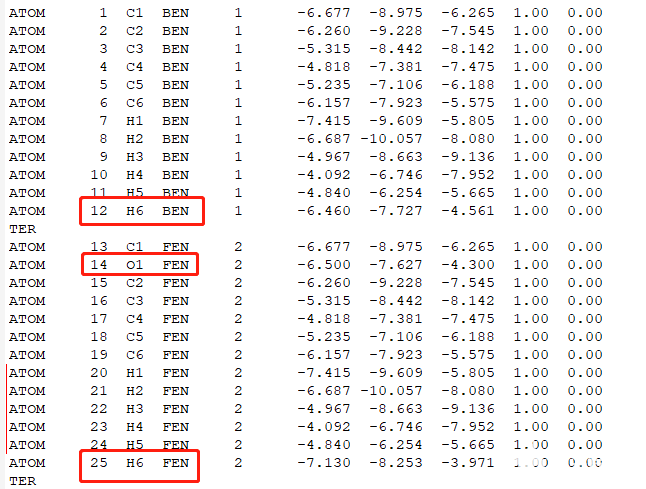
红框是有区别的原子,其他原子需要手动编辑使其保持一致的坐标,红线是编辑后的原子。
修改initial中的in文件下述部分。
timask1=':BEN',timask2=':FEN', scmask1=':BEN@H6',scmask2=':FEN@O1,H6'
使6个in文件符合实际更改的原子情况,只改变不同的原子,该步骤的目标是优化vdw变化过程中氢原子的位置。
使用sbatch run_v100.slurm,将任务提交到北鲲云的单卡V100集群上,该任务大概耗时1分钟,注意该任务如果有报错,可能是以下问题:
如果上述过程报关于ambmask的错(比如mask中不能用_符号),可以改写ambmask来获得正确可识别的mask。
ambmask-pcomplex_vdw_bonded.parm7-ccomplex_vdw_bonded.rst7-find:FEN
是ambmask的输入方式,在parm7和rst7中寻找这些原子,并用pdb的形式输出。
如果报错Error : Atom 12 does not have match in V1 ,说明这个原子在两个小分子配体中间的位置区别太大,TI不能识别这两个分子作为一样的背景,因此将这两个原子(初始和结束配体的)加入坐标中,就可以解决这个问题。
运行结束后,提取优化过后的分子结构。
pligands_vdw_bonded.rst7ligands_vdw_bonded.rst7.leap cppress_lig.rst7ligands_vdw_bonded.rst7 cpcomplex_vdw_bonded.rst7complex_vdw_bonded.rst7.leap cppress_com.rst7complex_vdw_bonded.rst7 cpptraj-pligands_vdw_bonded.parm7<<_EOF trajin ligands_vdw_bonded.rst7 strip ":1,2" outtraj ligands_solvated.pdb onlyframes 1 unstrip strip ":2-999999" outtraj ligands_BEN.pdb onlyframes 1 unstrip strip ":1,3-999999" outtraj ligands_FEN.pdb onlyframes 1 _EOF cpptraj -p complex_vdw_bonded.parm7 <<_EOF trajin complex_vdw_bonded.rst7 strip ":1,2" outtraj complex_solvated.pdb onlyframes 1 unstrip strip ":2-999999" outtraj complex_BEN.pdb onlyframes 1 unstrip strip ":1,3-999999" outtraj complex_FEN.pdb onlyframes 1 _EOF
再次使用tleap生成decharge,vdw和recharge的文件,decharge是配体1,recharge是配体2,修改阅读的pdb的名字即可。
tleap-f-<<_EOF
# load the AMBER force fields
source leaprc.protein.ff19SB
source leaprc.gaff2
source leaprc.water.tip3p
loadAmberParams frcmod.ionsjc_tip3p
loadOff BEN.lib
loadOff FEN.lib
loadamberparams BEN.gaff2.frcmod
loadamberparams FEN.gaff2.frcmod
# coordinates for solvated ligands as created previously by MD
lsolv = loadpdb ligands_solvated.pdb
lbnz = loadpdb ligands_BEN.pdb
lphn = loadpdb ligands_FEN.pdb
# coordinates for complex as created previously by MD
csolv = loadpdb complex_solvated.pdb
cbnz = loadpdb complex_BEN.pdb
cphn = loadpdb complex_FEN.pdb
# decharge transformation
decharge = combine {lbnz lbnz lsolv}
setbox decharge vdw
savepdb decharge ligands_decharge.pdb
saveamberparm decharge ligands_decharge.parm7 ligands_decharge.rst7
decharge = combine {cbnz cbnz csolv}
setbox decharge vdw
savepdb decharge complex_decharge.pdb
saveamberparm decharge complex_decharge.parm7 complex_decharge.rst7
# recharge transformation
recharge = combine {lphn lphn lsolv}
setbox recharge vdw
savepdb recharge ligands_recharge.pdb
saveamberparm recharge ligands_recharge.parm7 ligands_recharge.rst7
recharge = combine {cphn cphn csolv}
setbox recharge vdw
savepdb recharge complex_recharge.pdb
saveamberparm recharge complex_recharge.parm7 complex_recharge.rst7
quit
_EOF
生成好这些过程的文件后,使用setup_run.sh来产生三个步骤的输入文件,在修改setup_run.sh时,注意以下部分。
decharge_crg=":2@H6" vdw_crg=":1@H6|:2@O1,H6" recharge_crg=":1@O1,H6" decharge="ifsc=0,crgmask='$decharge_crg'," vdw_bonded="ifsc=1,scmask1=':1@H6',scmask2=':2@O1,H6',crgmask='$vdw_crg'" recharge="ifsc=0,crgmask='$recharge_crg',"
适配修改,注意H6的都改成初始的ambmask,O1,H6的都改成目标的ambmask,:前面的1或者2不要改。
如有必要修改λ,改变prod.tmpl和setup.sh中的值(0.00922开始的那一串)。
该文件将直接生成所需的slurm文件,并提交到对应的机器上,默认使用g-v100-1,运行pmemd.cuda,大致运行时间1小时左右。
有时候pmemd.cuda会运行失败,此时转用cpu来运行,使用run_mpi.py,提交命令:
pythonrun_mpi.pyligands500000mpi
将会检查所有ligands下的文件,对于5分钟内没更新,且info中运行步骤在500000 以下的,会提交到32核CPU机器上运行后续的模拟,直到结束。
这个运行步骤非常缓慢,使用32核CPU算完1纳秒的步骤可能需要7-8天,可以尝试先运行一段CPU,再运行一段GPU,改为python run_mpi.py ligands 500000 cuda即可。
运行结束后,使用alchemical-analysis/alchemical_analysis/alchemical_analysis.py来分析结果,注意该文件在python2下运行。
pip2installmatplotlib pip2installscipy pip2installnumpy pip2installpymbar==3.0.3
运行:
mkdir-pana_recharge&&cdana_recharge ../../alchemical-analysis/alchemical_analysis/alchemical_analysis.py-aAMBER-d.-p../recharge/[01]*/ti00[1-9]-qout-o.-t300-v-r5-ukcal-f50-g-w mkdir-p../ana_decharge&&cd../ana_decharge ../../alchemical-analysis/alchemical_analysis/alchemical_analysis.py-aAMBER-d.-p../decharge/[01]*/ti00[1-9]-qout-o.-t300-v-r5-ukcal-f50-g-w mkdir-p../ana_vdw&&cd../ana_vdw ../../alchemical-analysis/alchemical_analysis/alchemical_analysis.py-aAMBER-d.-p../vdw_bonded/[01]*/ti00[1-9]-qout-o.-t300-v-r5-ukcal-f50-g-w
此时只能输出三种变化的结果,将其TI 一列加和得到最终的结果。
如果见到pymbar的warning,只要注释掉对应的assert就可以了。
vim/home/cloudam/.local/lib/python2.7/site-packages/pymbar/timeseries.py
去第162行。

该应用场景解决将蛋白口袋内的残基A变为残基B所产生的相对自由能变。
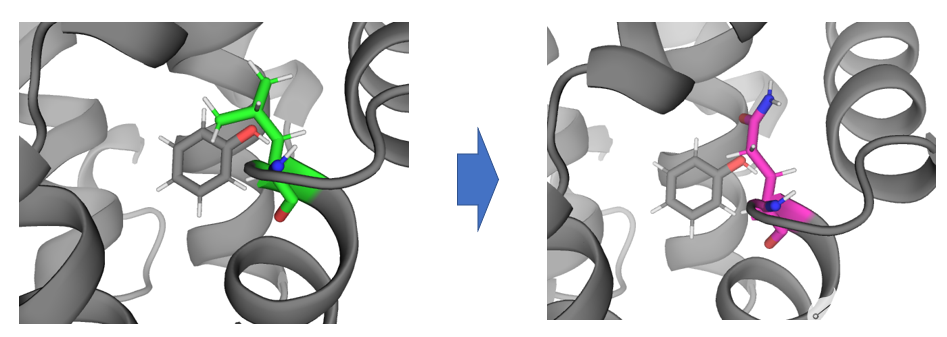
将蛋白口袋内的LEU转化GLN。
首先,使用pymol将分子打开,并选中残基,使用wizard-mutagenesis-protein完成突变,或者使用命令行完成突变:
load*.pdb
cmd.wizard("mutagenesis")
cmd.do("refresh_wizard")
cmd.get_wizard().set_mode("GLN")
cmd.get_wizard().do_select("86/")
cmd.get_wizard().apply()
cmd.set_wizard("done")
save*out_name.pdb,enabled
将突变后的蛋白文件保存为pdb文件。
将突变前的蛋白(WT.pdb)和突变后的蛋白(L86Q.pdb)的pdb文件上传。使用tleap,读取野生型结构。
tleap sourceleaprc.protein.ff14SB sourceleaprc.gaff2 sourceleaprc.water.tip3p loadamberparamsfrcmod.ionsjc_tip3p zn=loadpdbWT.pdb checkzn solvateBoxznTIP3PBOX10 addIonsznCl-0 savepdbznbox_check.pdb quit
记录盒子的范德华半径,并将结构中的水提取出来备用。
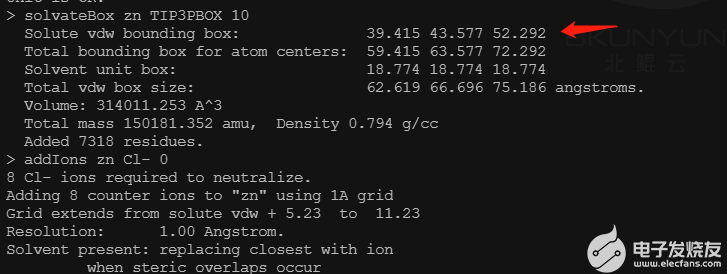
pythondry_for_TI.pybox_check.pdbwat.pdbWT_receptor.pdb
同样的方式读取突变型结构,使其保持Amber的原子顺序。
tleap sourceleaprc.protein.ff14SB sourceleaprc.gaff2 sourceleaprc.water.tip3p loadamberparamsfrcmod.ionsjc_tip3p zn=loadpdbL86Q.pdb checkzn solvateBoxznTIP3PBOX10 addIonsznCl-0 savepdbznL86Q_leap.pdb quit pythondry_for_TI.pyL86Q_leap.pdbwat1.pdbL86Q_dry.pdb
对比两个去水后的文件,发现由于Amber重编号,突变的残基变为84位,此时使用check_diff_online.py,来保证不是突变的残基的位置都绝对一致,以允许进行TI过程。
pythoncheck_diff_online.pyL84QL86Q_dry.pdbWT_receptor.pdb84L86Q_check.pdbWT_check.pdb
print的是空字典,说明所有的原子都是匹配的。
再次用tleap读取受体和配体(之前第一部分保存的mol2文件),并读取水盒子,其中ligand是刚才保存的配体(不变部分),m1和m2分别是突变前后的部分(注意突变只改变侧链,不改变主链),注意这里使用了刚才记录的范德华半径。
tleap
sourceleaprc.protein.ff14SB
sourceleaprc.gaff2
loadOffFEN.lib
loadamberparamsFEN.gaff2.frcmod
sourceleaprc.water.tip3p
loadamberparamsfrcmod.ionsjc_tip3p
ligand=loadmol2FEN.gaff2.mol2
m1=loadpdbL86Q_check.pdb
m2=loadpdbWT_check.pdb
w=loadpdbwat1.pdb
protein=combine{m1m2w}
complex=combine{m1m2ligandw}
setdefaultnocenteron
setBoxproteinvdw{39.41543.57752.292}
savepdbproteinprotein.pdb
saveamberparmproteinprotein.parm7protein.rst7
setBoxcomplexvdw{39.41543.57752.292}
savepdbcomplexcomplex.pdb
saveamberparmcomplexcomplex.parm7complex.rst7
quit
使用parmed处理protein.parm7和complex.parm7,以保证正确的所改变的位置提供给TI运算,此处的162为WT或者突变体的残基数,@后面的内容是python get_mutation.py L86Q得到的mapping结果,是在那个突变残基上但也没有变化的残基,84是突变位置,246是84+162。
parmedprotein.parm7<<_EOF loadRestrt protein.rst7 setOverwrite True tiMerge :1-162 :163-324 :84&!@CA,C,O,N,H,HA,CB :246&!@CA,C,O,N,H,HA,CB outparm merged_L86Q_protein.parm7 merged_L86Q_protein.rst7 quit _EOF parmed complex.parm7 <<_EOF loadRestrt complex.rst7 setOverwrite True tiMerge :1-162 :163-324 :84&!@CA,C,O,N,H,HA,CB :246&!@CA,C,O,N,H,HA,CB outparm merged_L86Q_complex.parm7 merged_L86Q_complex.rst7 quit _EOF
正确获得这些文件后,使用:
pythonauto_gene_inp_run.py84162CA,C,O,N,H,HA,CBL86Q
来生成tmpl文件(run.tmpl文件需要上传),并将tmpl文件转成真正的ti文件放进文件夹,同时使用slurm提交最小化,加热和运行步骤。
注意,这里直接使用cuda很容易断,可以适当自己修改之前的run_mpi.py来使用cpu续跑中断的模拟。
运行结束后的分析略。

本案例将实际运行一个蛋白-蛋白相互作用上的突变。我们计算新冠病毒受体结合区域(rbd)到人ACE2受体(ace2)复合物上发生Omicron的突变之一的返回突变A484E后的结合自由能变化。
生成连接了二硫键的大复合体水盒,记录vdw盒子大小,额外加入0.15M/L NaCL (总水数量*0.002772)。
tleap sourceleaprc.protein.ff14SB sourceleaprc.gaff sourceleaprc.water.tip3p loadamberparamsfrcmod.ionsjc_tip3p zn=loadpdbomi_SS.pdb bondzn.333.SGzn.358.SG bondzn.376.SGzn.429.SG bondzn.388.SGzn.522.SG bondzn.477.SGzn.485.SG bondzn.637.SGzn.645.SG bondzn.848.SGzn.865.SG bondzn.1034.SGzn.1046.SG checkzn solvateBoxznTIP3PBOX10 addIonsznNa+0 addIonsznNa+80 addIonsznCl-0 savepdbznbox_check.pdb quit pythondry_for_TI.pybox_check.pdbwat1.pdbomi_rbd.pdb,omi_ace2.pdb
同时生成一个小的水盒,用于跑蛋白部分的TI。
tleap sourceleaprc.protein.ff14SB sourceleaprc.gaff sourceleaprc.water.tip3p loadamberparamsfrcmod.ionsjc_tip3p m1=loadpdbomi_rbd.pdb bondm1.4.SGm1.29.SG bondm1.47.SGm1.100.SG bondm1.59.SGm1.193.SG bondm1.148.SGm1.156.SG checkm1 solvateBoxm1TIP3PBOX10 addIonsm1Na+0 addIonsm1Na+28 addIonsm1Cl-0 savepdbm1ligands_recharge.pdb quit pythondry_for_TI.pyligands_recharge.pdbrbd_wat.pdbtest_rbd.pdb
检查输入的是否在TI区域之外没有区别,注意输入的顺序,前面是突变后的蛋白,后面是原始的蛋白。
pythoncheck_diff_online.pyA152EA484E_rbd.pdbomi_rbd.pdb152A484E_check.pdbomi_check.pdb
设定正确的二硫键,分别加载两种水盒,输出protein和complex的拓扑学文件和坐标文件。
tleap
sourceleaprc.protein.ff14SB
sourceleaprc.gaff
sourceleaprc.water.tip3p
loadamberparamsfrcmod.ionsjc_tip3p
ligand=loadpdbomi_ace2.pdb
bondligand.308.SGligand.316.SG
bondligand.519.SGligand.536.SG
bondligand.705.SGligand.717.SG
m1=loadpdbomi_check.pdb
bondm1.4.SGm1.29.SG
bondm1.47.SGm1.100.SG
bondm1.59.SGm1.193.SG
bondm1.148.SGm1.156.SG
m2=loadpdbA484E_check.pdb
bondm2.4.SGm2.29.SG
bondm2.47.SGm2.100.SG
bondm2.59.SGm2.193.SG
bondm2.148.SGm2.156.SG
w1=loadpdbrbd_wat.pdb
w2=loadpdbwat1.pdb
protein=combine{m1m2w1}
complex=combine{m1m2ligandw2}
setdefaultnocenteron
setBoxproteinvdw{43.21553.42159.922}
savepdbproteinprotein.pdb
saveamberparmproteinprotein.parm7protein.rst7
setBoxcomplexvdw{64.17175.490114.587}
savepdbcomplexcomplex.pdb
saveamberparmcomplexcomplex.parm7complex.rst7
quit
使用parmed处理protein.parm7和complex.parm7,以保证正确的位置。
parmedprotein.parm7<<_EOF loadRestrt protein.rst7 setOverwrite True tiMerge :1-193 :194-386 :152&!@CA,C,O,N,H,HA,CB :345&!@CA,C,O,N,H,HA,CB outparm merged_A484E_protein.parm7 merged_A484E_protein.rst7 quit _EOF parmed complex.parm7 <<_EOF loadRestrt complex.rst7 setOverwrite True tiMerge :1-193 :194-386 :152&!@CA,C,O,N,H,HA,CB :345&!@CA,C,O,N,H,HA,CB outparm merged_A484E_complex.parm7 merged_A484E_complex.rst7 quit _EOF
正确获得这些文件后,使用:
pythonauto_gene_inp_run.py152193CA,C,O,N,H,HA,CBA484E
来生成tmpl文件(run.tmpl文件需要上传),并将tmpl文件转成真正的ti文件放进文件夹,同时使用slurm提交最小化,加热和运行步骤(5ns)。
审核编辑黄昊宇
发布评论请先 登录
相关推荐
超级电容器的划时代应用——新热力学第二定律
超级电容器的划时代应用——新热力学第二定律
超级电容器的划时代应用——新热力学第二定律
《新热力学第二定律极其系列专利试验、分析科研成果》的链接:发明家方面军
超级电容器的划时代应用——新热力学第二定律
《新热力学第二定律极其系列专利试验、分析科研成果》的链接:发明家方面军
链状分子流体混合物热力学性质的预测模型
聚合物溶液热力学模型的评述
浅析MCR框架的Web热力学数据库架构模式及其优势




 利用Amber热力学积分计算相对自由能变化
利用Amber热力学积分计算相对自由能变化











评论