全载玻片成像( WSI ),即使用全载玻片扫描仪对载玻片上的组织进行数字化,正在医疗保健领域获得广泛关注。 WSI 使组织病理学、免疫组织化学和细胞学方面的临床医生能够:
使用计算方法解释图像
将深度学习应用于数字图像以提高临床分析的准确性和再现性
提供有关患者数据的新见解
这篇文章解释了 GPU 加速工具包如何提高输入/输出( I / O )性能和图像处理任务。更具体地说,它详细介绍了如何:
使用 GPU 加速工具包将平铺数据从磁盘直接加载到 GPU 内存
使用 CUDA 执行图像处理
将处理后的磁贴写回磁盘
使用加速组织病理学技术节省时间对于快速识别和治疗疾病和疾病至关重要。
WSI I / O 和图像处理面临的挑战
将深度学习融入到整个幻灯片图像的处理中需要的不仅仅是训练和测试模型。使用深度学习的图像分析需要大量的预处理和后处理来改善解释和预测。整个幻灯片图像必须准备好用于建模,并且生成的预测需要额外的解释处理。示例包括伪影检测、颜色归一化、图像二次采样和去除错误预测。
此外,整个幻灯片图像的大小通常非常大,分辨率高于 100000 x 100000 像素。这将强制平铺图像,这意味着在建模中使用来自整个幻灯片图像的一系列子采样。将这些平铺图像从磁盘加载到内存中,然后处理平铺图像可能非常耗时。
加速 WSI I / O 和图像处理的工具
本文中介绍的用例使用 GPU 加速工具进行基准测试,具体如下。
cuCIM 公司
cuCIM ( C 计算 U 统一设备架构 C lara IM age ) 是一个用于多维图像的开源加速计算机视觉和图像处理软件库。用例包括生物医学、地理空间、材料和生命科学以及遥感。 cuCIM 库在许可证( Apache 2.0 )下公开可用,欢迎社区贡献。
Magnum IO GPU 直接存储
Magnum IO GPUDirect Storage (GDS) 提供存储 I / O 加速,这是 Magnum IO 库的一部分,用于并行、异步和智能数据中心 I / O 。 GDS 为 GPU 存储器和存储器之间的直接存储器访问( DMA )传输提供了直接数据路径,从而避免了通过 CPU 的缓冲区反弹。此直接路径增加了系统带宽,减少了延迟,并减少了 CPU 的使用负载。
总体而言, GDS 具有以下优势:
增加带宽,减少延迟,并减少数据传输的 CPU 和 GPU 负载
减少了性能影响和对 CPU 处理存储数据传输的依赖
对于完全迁移到 GPU 的计算管道,在计算优势之上充当力倍增器
支持与其他基于开放源代码的文件访问的互操作性,通过使用传统的文件 I / O (然后由使用 cuFile API 的程序访问),可以将数据传输到设备或从设备传输数据
GDS 可以通过 kvikIO RAPIDS 包从 Python 访问。 KvikIO 提供 Python 和 C ++ API ,能够在 GDS 支持下执行读或写操作。(当 GDS 不可用时,返回到基本 POSIX 和cudaMemcpy操作。)
整个幻灯片图像数据 I / O
当玻片在组织病理学中通过数字全玻片图像扫描仪数字化时,高分辨率图像将以多个放大倍数拍摄。 WSI 有一个金字塔形的数据结构,每个放大倍数的图像形成一个 WSI 的“层”。这些图像的最大放大倍数通常为 200 倍(使用放大倍数为 10 倍的 20 倍物镜)或 400 倍(使用 10 倍的 40 倍物镜)。
图 1 和图 2 显示了乳腺癌研究的基准用例比较。图 1 显示了 H & E 染色数字病理切片的放大视图,其中突出显示了感兴趣区域( ROI )。图 2 显示了与图 1 中突出显示的 ROI 相对应的高分辨率视图。
为此用例选择了具有以下特征的图像数据集:
数字图像中的层数为 XYZ
数字图像的最高分辨率层,包含 2028 个大小( 512 、 512 )的红、绿、蓝( RGB )色块
数字图像的总大小为 XYZ
数字病理学用例
本节介绍了三种不同的数字病理学用例。
用例 1 :将 WSI 瓦片加载到 GPU 数组中
在这个图像分析用例中,来自磁盘的 WSI 图像中的每个单独的区块都被加载到具有和不具有 GDS 的 1D GPU 缓冲区中。目标 GPU 阵列已预先分配。在每次使用中,读取区块都会被重新整形并放置在 GPU 输出阵列中的适当位置,如图 3 所示。
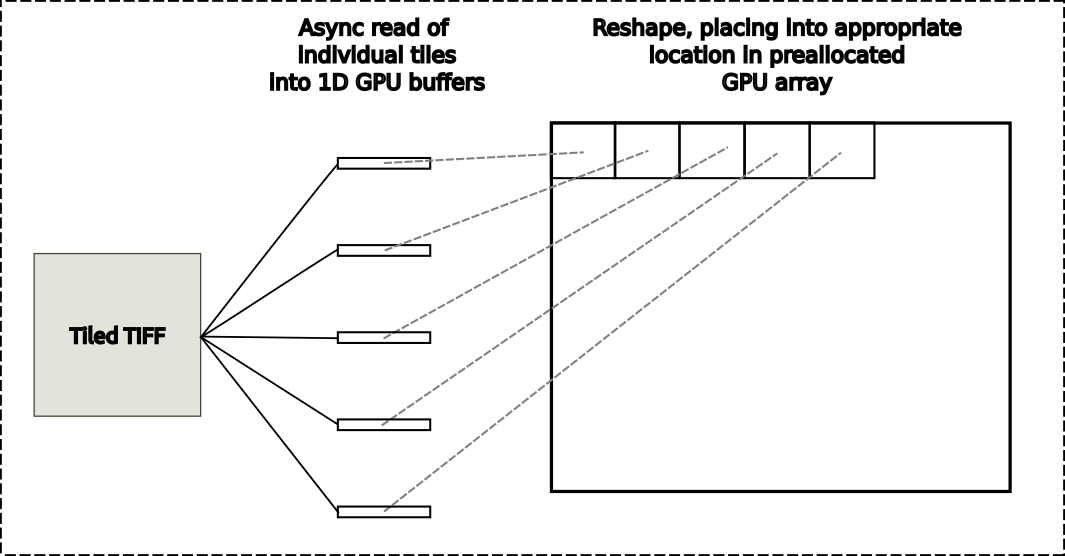 图 3 。从主机上的 平铺 TIFF 图像读取到 GPU 阵列
图 3 。从主机上的 平铺 TIFF 图像读取到 GPU 阵列
图 4 所示的基准测试结果绘制了与tifffile.imread的使用相关的加速度,然后调用cupy.asarray将阵列从主机传输到 GPU 。如果没有启用 GDS , kivikIO 为单线程和并行读取场景提供了 2.0 倍和 4.2 倍的加速。启用 GDS 后,单线程情况下的加速从 2.0 倍提高到 2.7 倍,而并行读取从 4.2 倍提高到 11.8 倍。
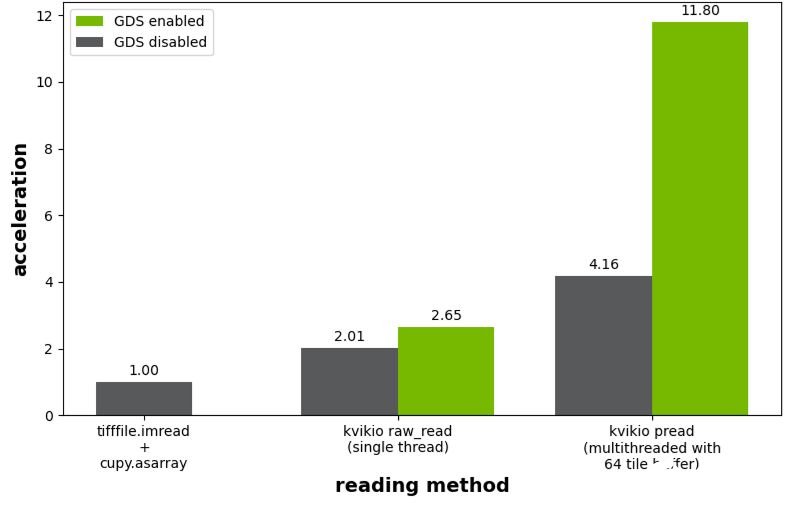 图 4 。相对于tifffile.imread和cupy.asarray的使用,平铺 TIFF 图像读取的性能
图 4 。相对于tifffile.imread和cupy.asarray的使用,平铺 TIFF 图像读取的性能
用例 2 :编写未压缩的 Zarr 文件
在此图像分析用例中,从 GPU 内存中的 CuPy 阵列开始。例如,您可以从用例 1 的输出开始。然后将此 GPU 数组平铺写入到单独的 Zarr 文件中,包括 GDS 和不包括 GDS 。这涉及到将 6084MB 的数据以每个“块”写入独立文件的格式写入磁盘。各种块形状的文件大小如下:
( 256 、 256 、 3 )== 768 kB
( 512 、 512 、 3 )== 3 MB
( 1024 、 1024 、 3 )== 12 MB
( 2048 、 2048 、 3 )== 48 MB
图 5 显示了块大小( 2048 、 2048 、 3 )的结果。对于除最小块大小( 256 、 256 、 3 )之外的所有块,启用 GDS (未示出)具有显著的好处。
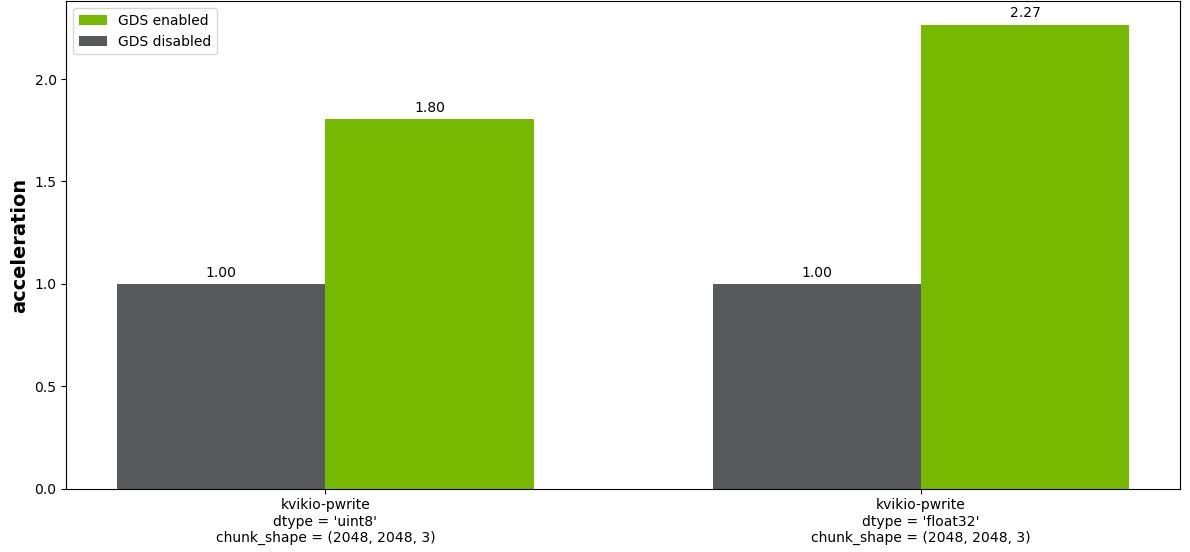 图 5. GDS 写入 Zar 文件(分块数组)的相对性能。显示了 8 位整数和 32 位浮点图像的结果。
图 5. GDS 写入 Zar 文件(分块数组)的相对性能。显示了 8 位整数和 32 位浮点图像的结果。
用例 3 :平铺图像处理工作流
此图像分析用例将加载、处理和保存整个幻灯片图像合并到一个应用程序中。这个用例如图 6 所示。步骤如下:
将单个平铺加载到 1D GPU 缓冲区
将平铺放置到预分配的 GPU 阵列的适当位置
使用基于 CUDA 的 Sobel 边缘检测内核的过程
将单个数据块写入 Zarr 文件到磁盘
一种方法是从用例 1 执行平铺读取,然后将 CUDA 内核应用于完整阵列,并从用例 2 执行平铺写入。这种方法具有相对较高的 GPU 内存开销,因为完整图像的两个副本必须存储在 GPU 内存中。在图 8 所示的基准测试结果中,这种方法被标记为“多线程全局”
一种内存效率更高的方法是并行异步读取、处理和写入各个区块。在这种情况下, GPU 内存需求大大减少,因为在任何一个时间只有一小部分图像在 GPU 内存中。这种方法如图 7 所示。在图 8 所示的基准测试结果中,这种方法被标记为“多线程平铺”
与禁用 GDS 的多线程全局方法相比,多线程平铺方法的结果在性能方面是标准化的。在没有 GDS 的情况下,执行异步平铺处理会有 15% 的性能损失,但 GDS 运行时提高了约 2 倍,比使用 GDS 的全局方法略快。
平铺 CUDA 处理方法的主要缺点是,由于平铺处理,简单实现目前无法处理潜在的边界伪影。这与像素计算无关,如颜色空间转换或数字病理学中从 RGB 到吸光度单位的转换。
然而,对于涉及卷积的操作,使用单个瓦片的边缘扩展而不是来自相邻瓦片的实际数据可能会产生细微的伪影。为了处理类似的图像分析用例,我们建议先保存到 Dask 数组,然后使用 map blocks 执行平铺处理,这可以考虑这些边界因素。
用于加速 WSI I / O 和图像处理的示例脚本
用于基准测试的脚本可以从 cuCIM repository 的 examples / python / gds _ whole _ slide 文件夹中获得。目前,这些图像分析用例使用 RAPIDS kvikIO 进行 GDS 加速读/写操作。这些组织病理学演示仅供说明,尚未进行生产使用测试。未来,我们希望扩展 cuCIM API ,为执行这些类型的平铺读写操作提供支持的方法。
总结:为什么加快 WSI I / O 至关重要
本文中介绍的图像分析用例表明, NVIDIA GPUDirect Storage 在减少需要读取和写入平铺数据集的各种高分辨率图像的 I / O 时间方面具有显著优势。使用加速组织病理学技术可以节省时间,这对于快速识别和治疗疾病和疾病至关重要。
-
NVIDIA
+关注
关注
14文章
4970浏览量
102957 -
gpu
+关注
关注
28文章
4722浏览量
128870 -
AI
+关注
关注
87文章
30642浏览量
268822
发布评论请先 登录
相关推荐
如何提高工程预算的准确性
合同智能审核软件-提高审查效率和准确性
讨论纹理分析在图像分类中的重要性及其在深度学习中使用纹理分析
什么是深度学习?使用FPGA进行深度学习的好处?
如何提高投标报价编制的准确性
AI可提高天气预报的准确性和准确性,助力农民和可再生能源行业
影响植物冠层图像分析仪准确性的因素是什么
如何将机器学习模型的准确性从80%提高到90%以上
应用深度学习分析提高基因组分析的准确性




 将深度学习应用于数字图像以提高临床分析的准确性和再现性
将深度学习应用于数字图像以提高临床分析的准确性和再现性











评论