单分子水平的蛋白质分析揭示了集合平均技术所掩盖的异质性行为。酶的数字量化通常涉及通过荧光底物的转换来观察和计算分成微区的单分子。然而,这种基于线性信号放大的策略仅限于周转率足够高的少数酶。
近期,来自法国巴黎物理化工学院 - 巴黎文理研究大学(ESPCI Paris-PSL Research University)的研究人员提出了一种基于分子编程和液滴微流控的替代性技术,用于对各种DNA和RNA加工酶进行数字检测。相关研究成果以“Functional analysis of single enzymes combining programmable molecular circuits with droplet-based microfluidics”为题发表在Nature Nanotechnology期刊上。
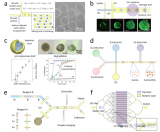
该研究利用一种被称为可编程超灵敏分子放大器(PUMA)的通用分子电路,该电路对DNA信号进行阈值指数放大,并与各种输入活动相耦合。PUMA包括一个转换模块,该模块将目标(此处为酶活性)与短DNA信号链的产生联系起来,还包括一个DNA-酶扩增系统,该系统最终会产生强烈的荧光读数。DNA-酶扩增系统由三个DNA模板组成:利用DNA聚合酶(Vent(exo-))和切口酶(Nb.BsmI)构成自动催化模板,实现催化信号链的指数复制;假模板使一部分信号链失活,从而起到催化引流的作用,以避免由渗漏反应引起的非特异性、非靶标依赖性扩增;而前荧光报告模板(rT)与信号链杂交,聚合后产生荧光信号。
此外,研究人员设计了一个转换模块,将扩增开关连接到用于诊断应用的缺口酶Nt.BstNBI(简称NBI)活性,以便用于诊断。研究人员在NBI浓度增加的情况下实时监控反应。正如预期的那样,目标浓度越高,信号链的产生越快,因此指数扩增开始得越快。该方法的整体灵敏度约为1μ/ml(每毫升毫单位,即约400 fM的活性酶),比基于荧光底物线性转化的分析低四个数量级。
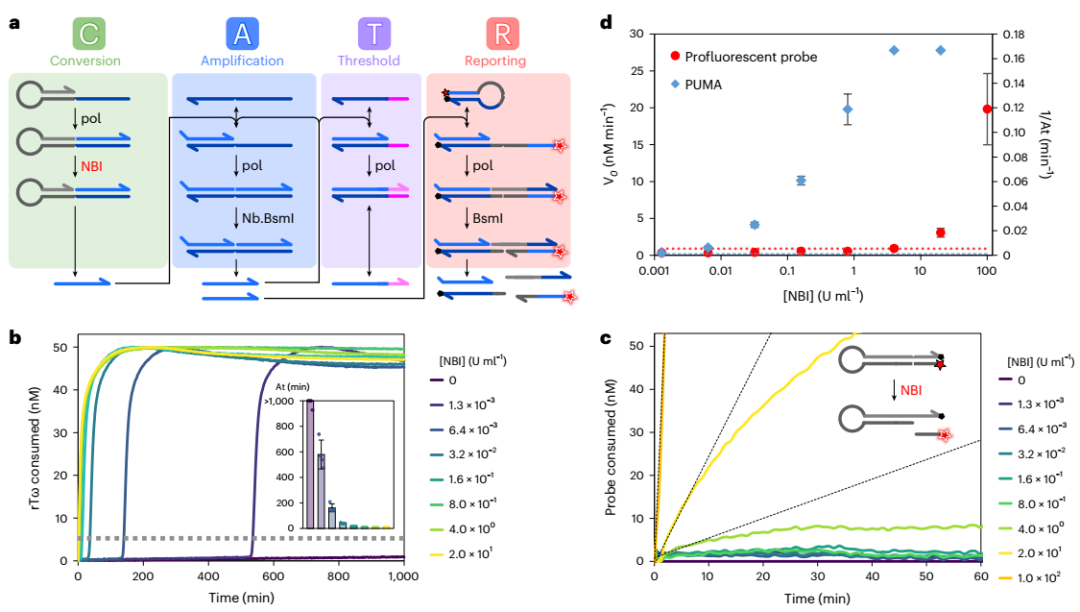
图1 NBI缺口酶活性的检测原理
利用基于PUMA的检测的高灵敏度特性,研究人员设想了一种通用方法,称为数字PUMA(dPUMA),用于单一酶的计数。为了对此进行评估,研究人员在NBI电路中加入不同浓度的目标酶,然后分成单分散的微滴,通过终点荧光显微镜对这些微滴进行扩增和分析。通过量化阳性液滴的比例并假设随机、泊松分布(方法),确定了活性酶的浓度,发现在广泛的稀释系列中,活性酶的浓度与初始输入浓度成比例,证实了该酶的准确数字检测。这种dPUMA工作流程可以与所有一锅法分析兼容,并适用于多种酶活性分子的绝对定量。引人注目的是,数字读数显示催化活性酶的浓度大大低于总酶浓度(由制造商提供),有时低几个数量级。在排除了可能的实验假象(如乳化过程中酶的失活或损失或大量污染蛋白的存在)后,研究人员得出结论,样品确实包含大量非功能性酶,这可能源于蛋白质生产、纯化和/或储存过程中的失活。
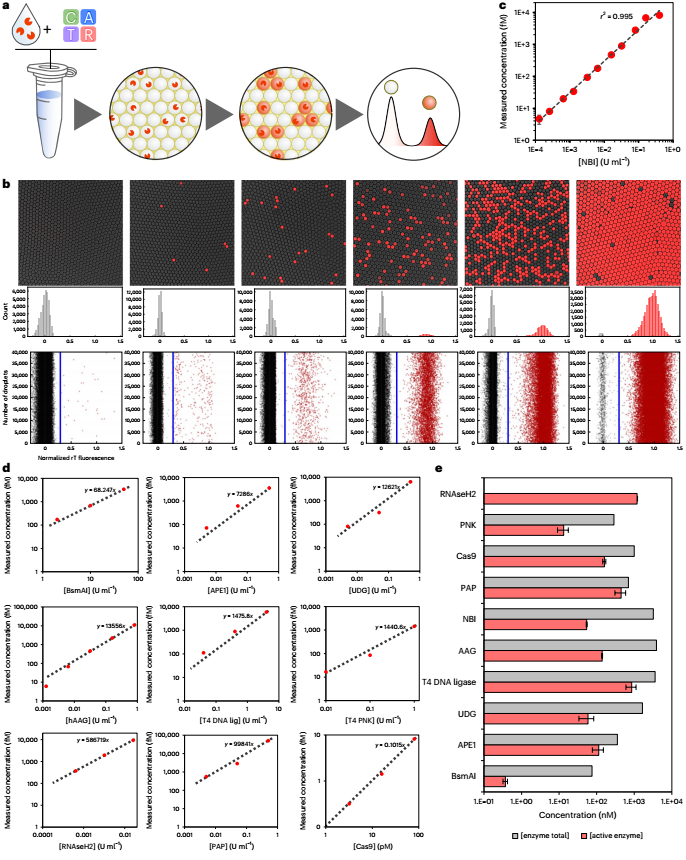
图2 DNA相关酶的数字检测
为了进一步研究酶混合物的组成和进化,研究人员使用实时版本的dPUMA方案评估了理论上相同的多肽库(商业NBI)的活性分布。研究人员乳化了不同酶浓度的样品,以测量不同泊松参数(λ)下的活性分布。每个被占据的液滴内的起始时间被用作酶活性的代表:酶活性越强,扩增越快,这一假设被批量测量中起始时间和酶活性之间的单调关系所证实。从图3f可以看出,在低λ(<2)下的扩增时间较窄,这使得区分不同占有率(一种、两种、三种或四种酶)的液滴尖峰成为可能。研究人员验证了活性分布由高斯函数的和进行了令人满意的拟合,这些高斯函数各自的权重受每个样本中λ测量值的泊松频率的约束。液滴占有率和泊松定律之间的一致性证明了通过数字分析测得的低活性分数不是由于酶的非泊松分布(这可能发生在聚集或结合到基因组DNA长片段的酶中)。重要的是,含有单一酶的液滴中的开始时间分布的变异系数仅为17%,这表明活性酶池具有相当均匀的活性分布。
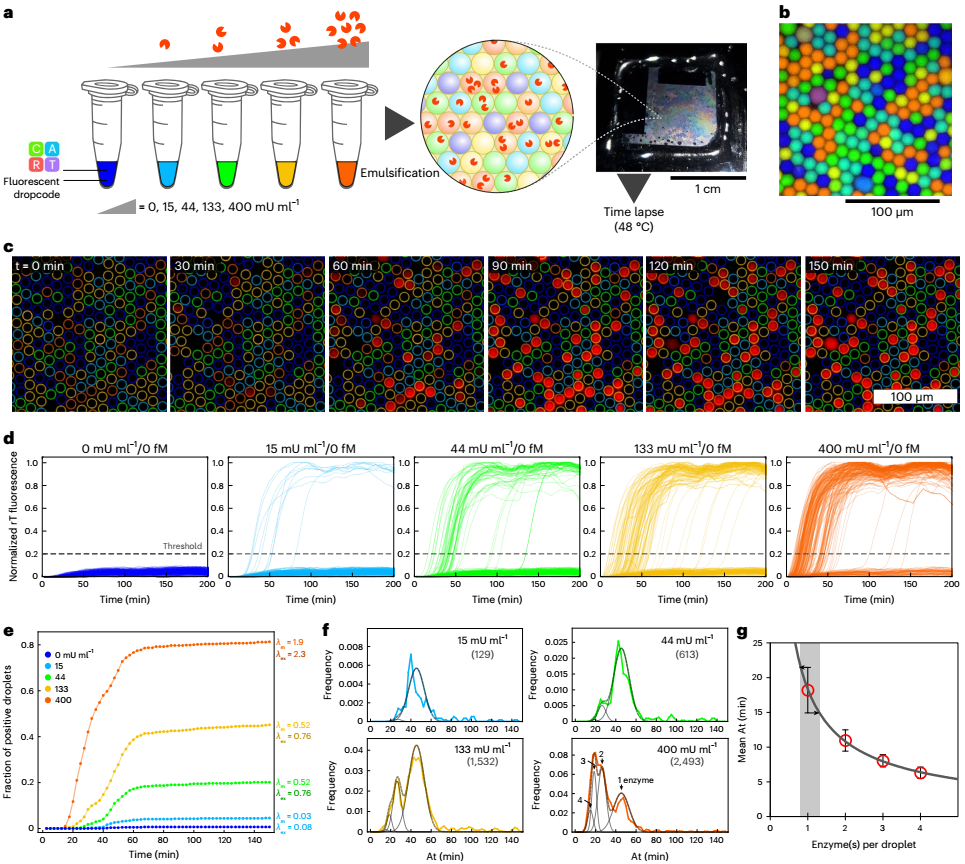
图3 通过延时实验评估多肽库活性分布
为了探索无活性组分的来源,研究人员评估了在各种应激条件下活性分布是如何受到影响的:包括热休克步骤的物理应激或化学应激。对于第一种情况,观察到随着热休克持续时间的增加,酶的活性部分呈指数衰减。然而,残留活性级分内的活性分布与未处理样品非常相似。无论热激温度如何,都可以观察到这种两种状态的行为,其中酶要么完全活性,要么完全失活。它与先前报告中描述的灾难性变性模型的概念相一致,在该模型中,加热的酶经历可逆的构象变化,直到达到临界点并不可逆地展开。
相反,在氧化处理过程中出现了一种明显的模式:随着H₂O₂浓度的增加,活性酶的比例再次下降;然而,开始时间的分布明显偏离了未经处理的样品:当氧化应激增强时,更多的单一酶表现出较低但可测量的活性。因此,轻度氧化应激导致一部分无活性的酶和一群具有中等活性的酶,这表明了一种机制,即活性降低的氧化底物在活性完全丧失之前是可接近的。活性位点外残基侧链的氧化可能会使催化袋不稳定,影响底物识别或降低支架的稳定性。在未处理的商业样品中没有观察到这种拓宽的活性分布,表明它们包含的全长多肽的无活性部分可能与折叠/聚集问题有关,而不是与化学损伤有关。
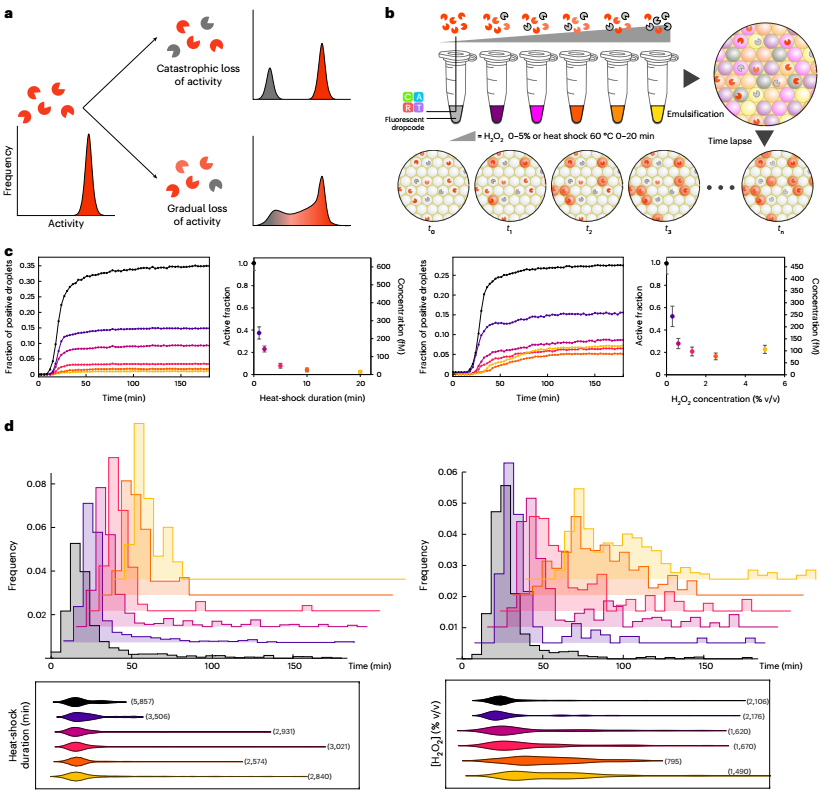
图4 酶群体的功能异质性研究
综上所述,研究人员通过将指数分子放大器的灵敏度与DNA酶电路和液滴读数的模块化相结合,可以在单分子水平上特异性地检测几乎所有与 D(R)NA相关的酶活性。这种被称为数字PUMA(可编程超灵敏分子放大器)的策略已在十多种不同的酶中得到验证,其中包括许多催化速率较慢的酶,甚至达到了化脓性链球菌Cas9表观单次周转的极限。数字计数法能独特地获得绝对摩尔定量,并能在所有测试的商业制剂中发现大量非活性催化剂。通过实时监测单个酶分子的放大反应,研究人员还提取了催化剂群体的活性分布,揭示了各种压力下的其他失活途径。该方法极大地扩展了可受益于单分子分辨率量化和功能分析的酶的数量。因此,数字PUMA 有望成为诊断或生物技术应用中精确酶定量的多功能框架。此外,这种数字测定方法还可用于研究蛋白质功能异质性的起源。
论文链接:
https://doi.org/10.1038/s41565-024-01617-1
-
放大器
+关注
关注
143文章
13596浏览量
213533
原文标题:基于分子编程和液滴微流控的单分子水平酶活性定量分析方法
文章出处:【微信号:Micro-Fluidics,微信公众号:微流控】欢迎添加关注!文章转载请注明出处。
发布评论请先 登录
相关推荐
NOVA无误差液滴微流体
基于轨道电润湿的液滴操控技术,有望用于新一代数字微流控平台





 一种基于分子编程和液滴微流控的替代性技术用于数字检测
一种基于分子编程和液滴微流控的替代性技术用于数字检测













评论