近日,天津大学精密仪器与光电子工程学院的光子芯片实验室提出了一种基于深度学习的二维拉曼光谱算法,成果以“Rapid and accurate bacteria identification through deep-learning-based two-dimensional Raman spectroscopy”为题,发表在《Analytica Chimica Acta》上。
表面增强拉曼光谱(Surface-enhanced Raman spectroscopy, SERS)可以提供分子独特的振动指纹,具有预处理简单、灵敏度高和无创检测等优点。随着人工智能(artificial intelligence, AI)技术的快速发展,基于AI的拉曼光谱分析在细菌识别等生物传感领域具有广阔前景。目前,为了更加精确地实现细菌识别,研究者们提出将一维拉曼光谱转换为二维拉曼光谱图来获取更加丰富的数据信息。但是,高分辨率的二维拉曼光谱图在提高识别准确度的同时,通常会引起计算时间过长等问题。
在本项工作中,研究者们提出了一种低分辨率、多信息量的二维拉曼光谱图处理算法,验证了其在细菌识别中的可行性。该算法基于小波包变换与格拉姆角场(Wavelet Packet transform and Gramian Angular field, WPGA)技术,利用深度学习模型实现快速高效地识别细菌。如图1所示,识别过程主要包括三个步骤,首先,采集细菌样品的SERS光谱并进行预处理;其次,利用WPGA算法将一维拉曼光谱转换成二维拉曼光谱图,这是本项工作能够实现快速高效分类的关键。由于分子振动,拉曼光谱常会出现微小的特征峰,但由于这些信息频率相对较高,在光谱平滑过程中容易被忽略。因此,我们采用小波包变换技术,对高频信息成分进行更为精细的筛选,以减少有用信息的损失。然后利用格拉姆角场技术,对筛选后的有效特征添加序列信息来提高识别精确度。最后,通过训练深度学习模型来识别细菌。
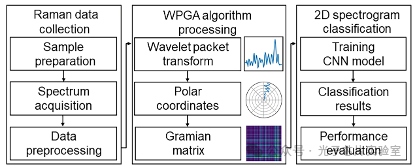
图1.细菌识别的主要步骤。包括三个步骤:拉曼数据收集、WPGA算法处理和二维光谱图分类。
基于提出的算法,研究者们对枯草芽孢杆菌和酵母菌的SERS光谱进行了分类验证。图2(a)展示了基于卷积神经网络进行该二元分类问题的训练过程,结果表明验证集的精度可以达到99.75%,损失为0.0074。图2(b)展示了模型训练完成后,使用测试集来评估分类性能,识别准确率为99.64%,初步证明了该算法的可行性。为了验证该算法的泛化能力,研究者们进一步开展了基于Bacteria-ID公共数据集的多分类识别任务。图2(c)展示了没有进行WPGA算法处理时,30种细菌在二维空间中的分布,不同类别之间的相互混合,难以区分。如图2(d)所示,利用所提算法后,不同种类的细菌彼此分离,基于Resnet网络模型的分类识别准确率可以达到90.55%。
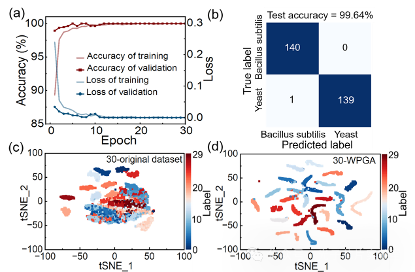
图2.两种及多种细菌的分类识别结果。(a)训练集和验证集的准确率和损失曲线;(b)测试集的混淆矩阵;(c)数据集在二维空间中的分布;(d)使用WPGA算法后,数据集在二维空间中的聚类效果。
本论文第一作者为天津大学精密仪器与光电子工程学院的硕士生刘怡辰,通信作者为天津大学精密仪器与光电子工程学院的程振洲教授、胡浩丰教授和高翊盛博士。该工作得到了国家自然科学基金(62161160335、62175179、62475188、61805175、12304428)、天津市自然科学基金(23JCJQJC00250)、广东省自然科学基金(2022B1515130002)和日本学术振兴会(JP18K13798)项目的支持。
-
算法
+关注
关注
23文章
4587浏览量
92480 -
二维
+关注
关注
0文章
39浏览量
11968 -
深度学习
+关注
关注
73文章
5463浏览量
120875 -
拉曼光谱
+关注
关注
0文章
82浏览量
2716
原文标题:面向细菌识别应用的二维拉曼光谱处理算法
文章出处:【微信号:光子芯片实验室,微信公众号:光子芯片实验室】欢迎添加关注!文章转载请注明出处。
发布评论请先 登录
相关推荐
一种基于二维离散小波变换的医学图像增强算法
广义二维相关光谱学发展
一种快速二维到来方向估计算法
基于深度学习的二维人体姿态估计算法

从单层石墨烯中收集拉曼光谱





 一种基于深度学习的二维拉曼光谱算法
一种基于深度学习的二维拉曼光谱算法















评论