自2009年汤富酬研究员首次报道其开创性工作以来,单细胞RNA测序技术已经逐渐成为生物医学研究的重要方法,其在发育生物学和干细胞领域展现出强大的应用前景。随着单细胞转录组扩增方法的不断优化,以及测序通量的持续提升,研究人员已经不再满足于几十甚至几百个单细胞所提供的信息量。
2015年以来,一系列基于细胞条形码标记的高通量单细胞RNA测序技术被相继报道,其中尤以哈佛大学David Weitz实验室开发的inDrop和Drop-seq,以及10X Genomics公司开发的Chromium系统最受关注。然而,如何从这些技术中选择出最适合于自己项目的方法,成为每一个想从事单细胞转录组分析的科研人员首先面对的问题。
2018年11月21日,清华大学生命学院王建斌研究组和北京大学BIOPIC黄岩谊研究组以及上海交通大学附属瑞金医院刘峰研究员紧密合作,在Molecular Cell上发表了题为Comparative analysis of droplet-based ultra-high-throughput single-cell RNA-seq systems的研究论文,系统比较了inDrop、Drop-seq和Chromium三个系统在单细胞RNA测序方面的表现,并初步尝试了基于inDrop系统的个性化开发,为未来研究中合理选择检测平台提供了数据支持。
液滴超高通量单细胞RNA测序系统(droplet-based ultra-high-throughput single-cell RNA-seq systems)基于一个相同的原理,即利用微液滴将单个细胞与单个微球配对包裹,每个微球上带有的独特寡聚核苷酸序列可以对来自于不同单细胞的RNA进行区分标记,之后就可以对数千个单细胞的cDNA进行合并操作,每一条cDNA的细胞来源可以在后续的生物信息学分析中进行解读。此方法使得实验人员不必针对每一个单细胞进行繁琐的重复操作,大大提高了实验效率。
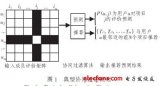
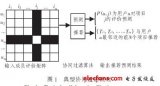
研究人员首先系统比较了三个系统的原理设计(下图),尤其是微球及寡聚核苷酸序列的特性。通常情况下,细胞和微球需要分别经过高倍稀释,从而避免同一液滴内存在多个细胞或者微球。由此导致的双泊松分布大大降低了细胞的包裹效率。inDrop和Chromium系统所使用的大尺寸水凝胶微球可以被挤压通过狭窄的微流通道,使得绝大部分液滴中有且仅有一个微球,细胞的利用率得到了显著的提高。另一方面,inDrop系统所使用的CEL-seq单细胞转录组扩增技术耗时较长,整个实验无法在当天完成。

为了有效比较三个系统的技术参数,研究人员选择使用均质的淋巴母细胞作为实验材料,并开发了一套同时兼容三个平台数据的生物信息学分析流程。在数据分析过程中,研究人员首先发现微球表面的寡聚核苷酸序列并不完美,尤其是inDrop和Drop-seq系统所用微球,普遍存在内部序列不均一的问题。即使经过序列校正,inDrop和Drop-seq仍有约一半的测序数据无法被有效归入具体的细胞,导致的测序数据的浪费和检测灵敏度的下降。另一方面,UMI(unique molecular identifier)的加入可以有效提高inDrop和Drop-seq数据的均一性,而其对Chromium数据的改善非常有限。最终,在基因数和转录本数方面,三个系统所产生的数据均可以用于细胞亚型分析,其中Chromium系统展示出相对更高的灵敏度。
除了常规的技术参数分析以外,研究人员在对比三个系统所产生数据时还发现了显著的系统特有差异,具体表现为数据按系统来源显著分成了三个亚群,这提示大家未来在比较不同系统来源的数据时,要首先注意系统导致的表达差异。此外,针对inDrop系统特有的开发性,研究人员尝试将单细胞转录组扩增方式由CEL-seq改为Smart-seq,并获得了初步的成果,这展示出inDrop系统在特殊应用领域的潜在优势。

近年来,针对整个器官乃至整个生物个体的超大规模单细胞研究项目逐渐成为国际会议中讨论的焦点,其中尤以美国和欧洲科学家联合开展的人类细胞图谱(Human Cell Atlas)计划最为著名。希望本文能够对有意开展单细胞转录组研究的科研人员提供有效的指导。
据悉,清华大学生命学院王建斌研究员和北京大学BIOPIC黄岩谊教授为本文通讯作者,北京大学BIOPIC张先念博士、清华大学生命学院博士生李天奇、上海交通大学附属瑞金医院刘峰研究员、北京大学深圳研究生院博士生陈雅琪为本文共同第一作者。
-
RNA
+关注
关注
0文章
46浏览量
9719 -
数据分析
+关注
关注
2文章
1452浏览量
34076
原文标题:北京大学黄岩谊教授:比较液滴超高通量单细胞RNA测序
文章出处:【微信号:Microfluidics-Tech,微信公众号:微流控科技】欢迎添加关注!文章转载请注明出处。
发布评论请先 登录
相关推荐
家长汇个性化测评系统的创新型招生策略(二)
HarmonyOS SDK,赋能开发者实现更具象、个性化开发诉求
基于Web挖掘的个性化网络学习系统设计
Mascom已经选择Bercut为其提供个性化彩铃解决方案
知识个性化推荐系统研究

AI、大数据、脑科学个性化学习系统大讲解
利用Pocket IO™ PLC开发平台展示未来工厂
大规模个性化定制研究综述!





 基于inDrop系统的个性化开发,为未来研究中合理选择检测平台提供了数据支持
基于inDrop系统的个性化开发,为未来研究中合理选择检测平台提供了数据支持












评论