医学图像是临床诊断主要依据之一,对医学图像的处理分析具有十分重要的理论和临床价值。本文详细分析了医学图像处理面临的挑战,阐述了医学图像传统处理流程以及GPU的作用,总结了人工智能医学影像发展现状、问题以及GPU算力加持下深度学习医学图像处理算法的演进趋势。
医学图像处理面临的挑战
自伦琴1895年开创X射线人体成像先河以来,计算机断层扫描 (CT) 、磁共振成像 (MRI) 、正电子发射断层成像 (PET) 等医学影像技术已然成为临床医生最直接、最常用的方法手段,它在整个临床应用(包括疾病诊断、手术治疗、预后评估等)中起着至关重要的作用。医学图像的处理分析是医学影像技术的核心部分,包括图像重建、增强、配准、融合、跟踪、分割、识别、可视化等技术,不仅可以辅助医生阅片分析、自动化诊疗过程,还是手术机器人等图像引导介入 (Image Guided Interventions,IGI) 手术的关键技术。
然而,医学图像处理面临诸多挑战。
1、随着各类医学影像设备的迅速发展和普及,医学图像数据正以前所未有的速度增加。据统计,美国的医学图像数据年增长率为63.1%,我国图像数据年增长率也在30%左右。截止2020年,全球医学图像数据量已经达到了惊人的40万亿GB[1]。
2、如图1所示,不同类型的成像设备,由于成像原理不同,其生成的不同模态图像在相同解剖结构位置上并不存在简单的线性对应关系。在临床应用中,医生往往需要结合多种模态图像进行融合分析、互为补充,以改善临床诊断、病情监测、外科手术引导、疗效评估的水平。
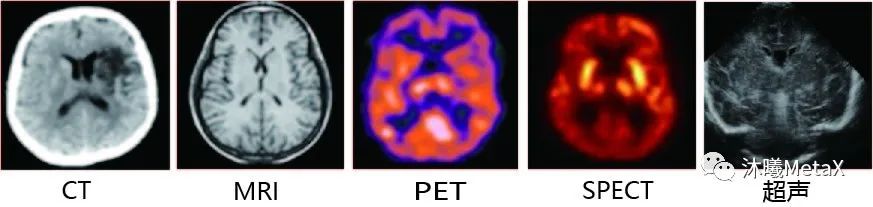
图1:不同模态的脑部图像
来源:F. E.-Z. A. El-Gamal, M. Elmogy, A. Atwan. Current trends in medical image registration and fusion. Egyptian Informatics Journal, 2016
3、医学图像个体差异较大,即使是同一台设备同一病人,由于成像系统的缺陷,不同成像参数条件和患者呼吸运动等多种原因,也容易造成医学图像出现不同程度的伪影和噪声。
4、医学图像的解释并没有完全统一的标准。由于经验和环境条件不一样,即使是同一医生在不同时间段对同一张图片都有可能出现不同的解释和描述。
5、临床诊疗对时间具有一定敏感性。对于一些实时性要求较高的临床应用,如手术导航,图像处理速度要求至少达到 25-30帧/秒。
医学图像的这些特点,对医学图像处理的算法和算力都提出了较高的要求。近年来,随着人工智能 (artificial intelligence,AI) 核心——深度学习在计算机视觉处理领域的巨大成功,AI技术通过替代或增强医学图像处理的部分或整体流程,可以显著提高医学图像分析的精度和效率,提升健康与诊疗的效益及价值。GPU具有高效的并行计算能力,不仅适合计算密集型传统图像处理任务,而且是人工智能模型训练、预测的关键硬件。接下来,我们将分别对GPU的作用进行阐述。
GPU提速医学图像处理全流程
医疗成像设备产生的医学图像具有多种维度,包括2D、3D、4D图像,以及2D视频图像(内窥镜或显微镜视频)。随着图像维度和算法复杂度的提高,医学图像处理的计算量也在大幅增加。对于大多数医学图像处理算法,图像像素可以作为线程进行独立处理,具有数据并行计算的特点,这是GPU并行加速的基础。GPU具有高并行、多线程、多流式处理器、高内存带宽的特点,可以实现医学图像处理全流程的高性能加速计算。图2给出了医学图像处理的基本流程,下文将分别予以介绍。
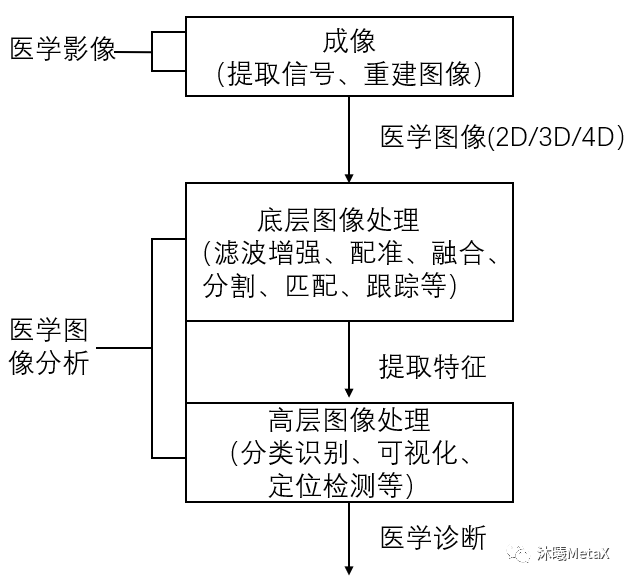
图2:医学图像处理基本流程
图像重建
图像重建是指利用传感器或能量源测量人体数据,经重建算法获得人体三维结构或功能信息。图像重建是医学图像处理中较为耗时的过程,为了缓解计算压力,研究者过去常使用较为简单的成像模型进行直接解析。例如,在CT重建中,FBP (Filtered back projection,滤波反投影) 是解析法中较为常见的算法。在FBP中,将投影数据进行FFT滤波处理后,利用系统权重矩阵和投影数据的相乘沿投影方向重建三维图像。CT投影重建模型如图3所示,探测器接收的信号为X射线管源穿过人体的X射线投影数据。为获得CT完整断层图像,需要X射线沿人体中心进行至少180°的连续旋转。
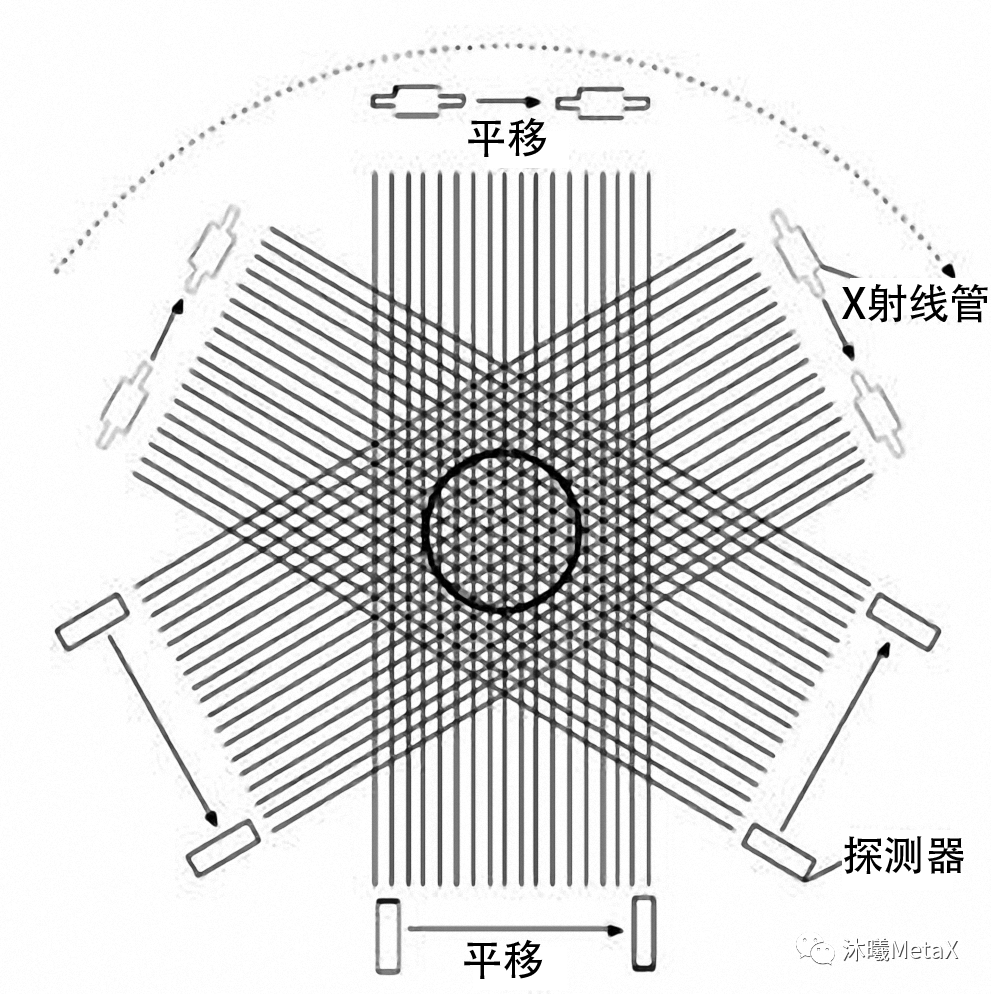
图3:CT投影重建模型
尽管解析算法简单、重建速度快,但由于未引入真实的成像过程,如未考虑噪声等,其成像质量较差,且要求较高信噪比的测量数据,但这往往受限于放射剂量或系统约束。近年来,迭代法被广泛用于图像重建过程中,可以显著减少放射剂量、降低噪声、提高成像质量。表1总结了几种常用医学成像技术的重建原理及相关技术特点。
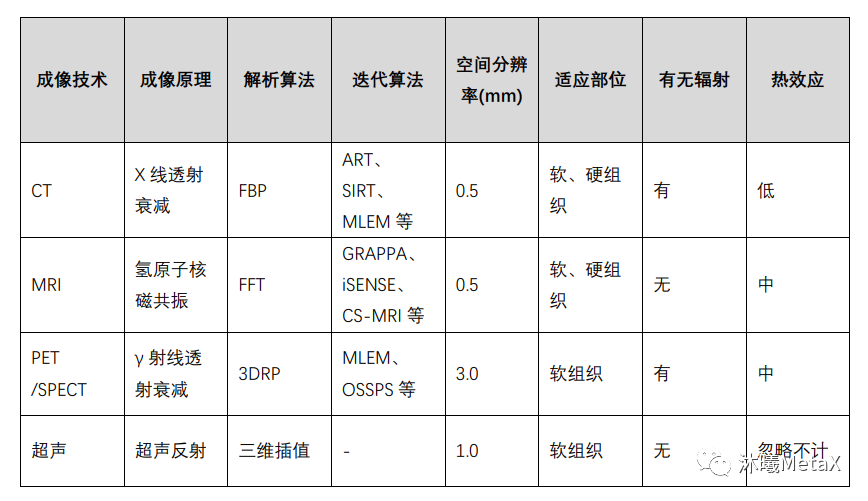
表1:几种常用医学成像技术对比
迭代重建算法的基本原理是估计初始三维图像,对其进行前向成像模拟,并与实际测量数据进行比较,根据误差结果校正三维图像估计,重复该过程直至收敛。这一过程中,需要大量的数据计算。以往,图像重建常使用ASIC (Application Specific Integrated Circuit,专用集成电路) 、FPGA (Field Programmable Gate Array,现场可编程门阵列) 、DSP (Digital Signal Processing,数字信号处理芯片) 等芯片进行加速。相比这些芯片,GPU可以提供更好的编程灵活性和更合理的开发成本性能比。目前,在图像重建领域有大量使用GPU加速的开源软件和医疗设备厂商商用软件。通过对重建过程任务并行化、内存合并优化以及减少线程发散和竞争等手段,GPU的加速性能可以显著提高。表2给出了MRI重建CS迭代方法的各模块的GPU加速比,在GPU的加持下,其总加速比可以达到53.9倍[2]。
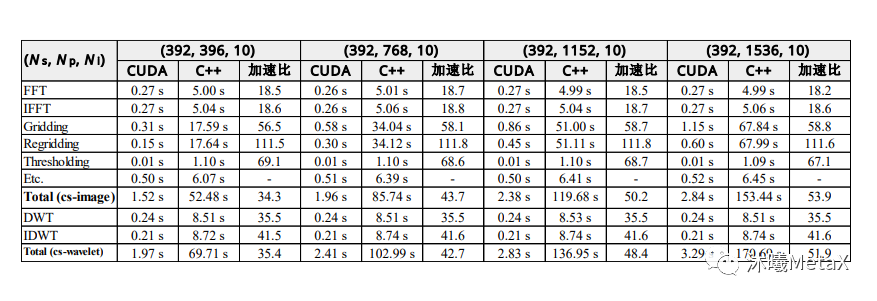
表2:CS-MRI重建中两种方法的GPU加速比较。
表中给出了执行单次迭代的各模块平均时间和加速比。
其中,Ns为采样数,Np为投影数量,Ni为轨迹数。
来源:S. Nam, M. Akçakaya, T. Basha, C. Stehning, W. J. Manning, V. Tarokh, et al. Compressed sensing reconstruction for whole‐heart imaging with 3D radial trajectories: a graphics processing unit implementation. Magnetic resonance in medicine, 2013, 69(1): 91-102
滤波增强
原始医学图像受成像设备和获取条件等多种因素的影响,易受到噪声的污染,会出现图像质量的退化或畸变。对医学图像进行滤波增强,可以抑制噪声、减少失真、增强图像信息,以便后续图像处理分析。传统的图像滤波增强技术分为空间域和频域两种,两者都可以用来消除图像噪声,并且同时达到增强图像特征的目的。常用中值滤波、高斯滤波、扩散滤波和双边滤波等方法进行处理。这些滤波算法等价于图像域的可分解卷积运算,或者傅里叶频域空间的乘积运算,因此极其适合GPU并行加速计算。表3给出了对三维图像(512x512x128)进行不同参数非局部均值去噪加速效果比较,其GPU加速比平均可以达到30多倍[3]。
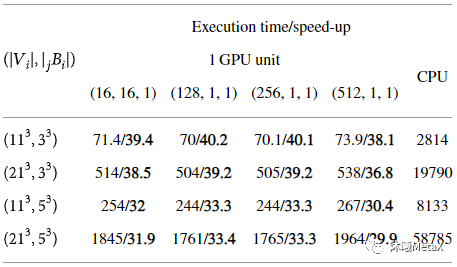
表3:不同参数三维非局部均值去噪加速效果比较
(执行时间(s)/加速比)
来源:S. Cuomo, P. De Michele, F. Piccialli. 3D Data Denoising via Nonlocal Means Filter by Using Parallel GPU Strategies. Computational and Mathematical Methods in Medicine, 2014
图像配准
医学图像配准是指对同一人的同一解剖部位,利用某些意义不同的成像手段形成的意义不同的两幅影像,寻求一种(或一系列)空间变换,使这两幅医学影像的解剖结构对应点达到空间位置上的尽可能一致。这里的一致是指人体上同一解剖点或者至少是所有具有诊断意义的点以及手术感兴趣点都达到匹配。医学图像配准是图像分割、图像融合等后续图像处理的基础。
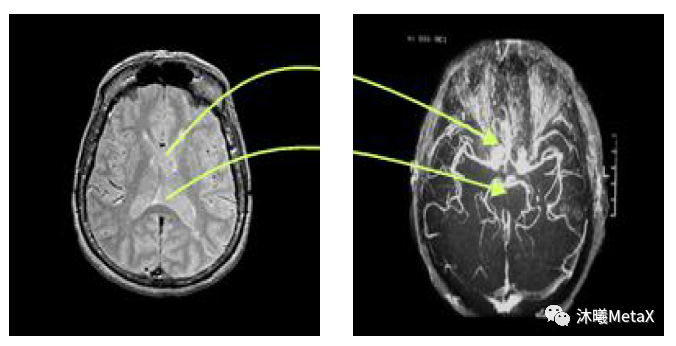
图4:大脑 MRI和 DSA图像配准示意图
如图5所示,图像配准过程中,作为配准目标而不发生变化的待配准图像称为参考图像R,另一幅要进行空间变换的待配准图像称为浮动图像M,整个配准过程可以看作是对浮动图像M进行有限次几何变换T后,使其变换后图像同参考图像R相似度无限接近的过程。
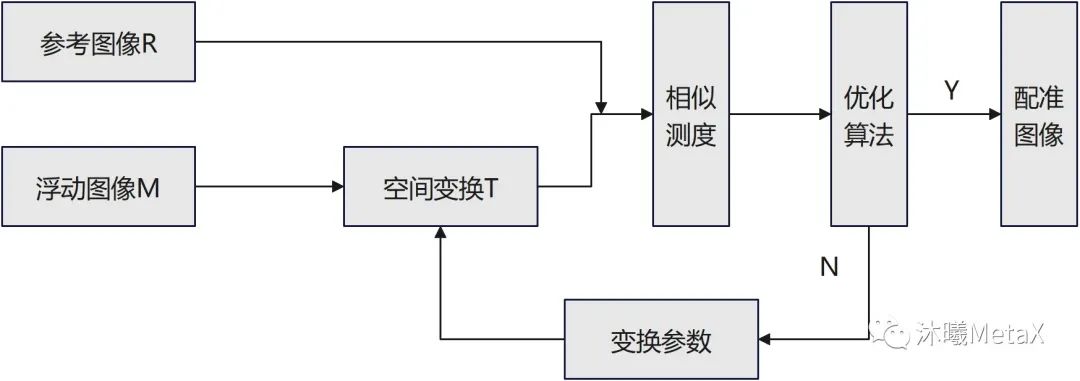
图5:医学图像配准框架示意图
医学图像配准方法的核心包括三个部分:空间变换、相似测度、优化算法。空间变换主要涉及像素空间的矩阵和插值运算,GPU对矩阵和插值运算有内置加速设置;相似性测度则是利用图像灰度或特征信息,量化图像间的匹配相似程度,利用并行、合并算法, 可以实现GPU的加速;而优化算法主要为顺序过程,较难实现相关GPU加速。表4给出了一种三维弹性配准GPU加速情况,可以看出尽管优化算法无法实现并行加速,GPU整体加速比仍能达到4.69倍[4]。
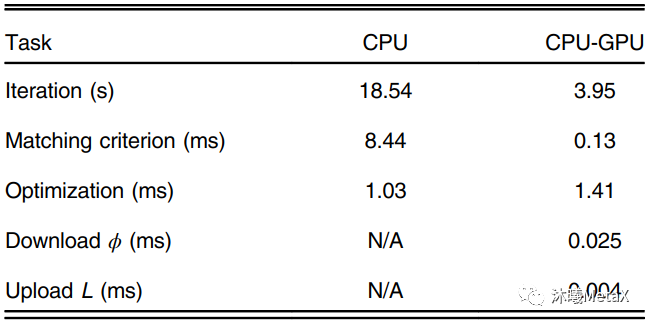
表4:一种三维弹性配准的各模块GPU加速情况
来源:S. Ekström, M. Pilia, J. Kullberg, H. Ahlström, R. Strand, F. J. J. o. M. I. Malmberg. Faster dense deformable image registration by utilizing both CPU and GPU. 2021
图像匹配
图像匹配和图像配准近似,目的是寻找与一幅图相似的图像,但无需对寻找到的图像做空间矫正。根据匹配时利用的信息不同,可将匹配算法分为三个类别,分别是:基于特征描述符的匹配方法、基于空间位置信息的匹配方法和融合了特征信息和位置信息的匹配方法。根据得到的匹配数量的多少,又可将匹配算法分为稀疏匹配算法、准稠密匹配算法和稠密匹配算法。图像匹配在医学图像领域中被广泛用于基于内容的图像检索 (Content Based Image Retrieval,CBIR) ,通过从现有医学图像数据库中快速查询图像相似的临床医学图像,医生可以参考病理相似病例的诊断方法以辅助其作出正确的临床诊断。
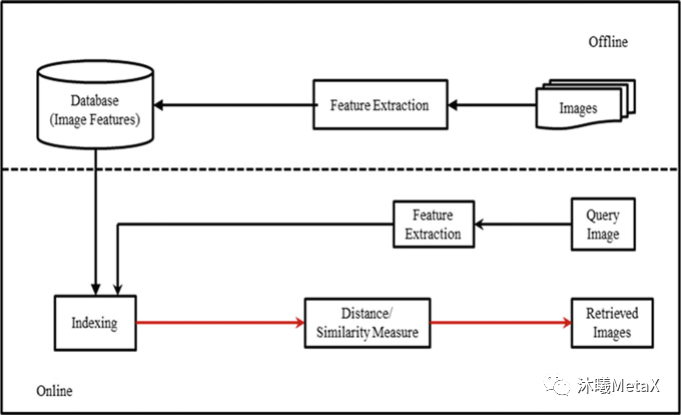
图6:基于内容的图像检索系统基本流程
来源:L. R. Nair, K. Subramaniam, G. K. D. Prasannavenkatesan. A Review on Multiple Approaches to Medical Image Retrieval System. in: Intelligent Computing in Engineering. Singapore, 2020
如图6所示,CBIR过程通常分为特征提取、特征索引、相似性度量等过程。这些过程都可以利用GPU进行并行加速。一项研究表明[5],利用GPU可以实现4~5倍的加速。
图像融合
正如前文所述,不同类型的医学影像设备为患者临床诊疗提供了多层面、多角度、多节点的图像数据,不同模态的数据间既存在冗余信息,又充斥着大量的互补信息。多模态医学图像融合充分利用不同模态图像的互补性和冗余性,对同一病灶部位不同模态的医学影像进行融合,可以得到比原图像信息更丰富、更精准的融合图像。通常,多模态医学图像融合首先需要对不同模态的图像进行配准,使得同一解剖部位的像素位置在不同图像下能够一一对应,之后根据设定的策略融合各模态中的有用信息,生成质量更好的图像。如图7所示,根据图像数据处理层次的不同,图像融合主要分为像素级融合、特征级融合和决策级融合。
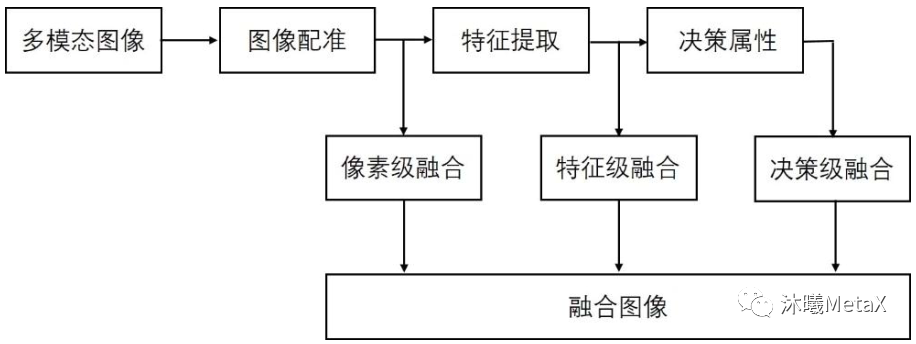
图7:不同层次图像融合系统示意图
其中,像素级多尺度变换方法是目前图像融合算法的主流,在图像精确配准前提下,对两幅或多幅原图像使用分解变换方法分别进行分解,得到不同频率层次的图像,然后将两幅图像对应的低频系数和高频系数分别进行融合,获得融合系数,最后将融合后的系数进行傅里叶逆变换,得到最终的融合图像。利用GPU可以加速傅里叶变换等过程,从而实现近10倍的加速[6]。
图像分割
图像分割,是指将图像分成若干不相互重叠的子区域,使得同一个子区域内的像素特征具有一定的相似性、不同子区域之间像素特征呈现较为明显的差异,常用于分割脑部区域、血管、器官、肿瘤、骨骼等,是许多基于医学图像的诊断、治疗和分析应用的重要基础步骤。
常用的传统图像分割方法包括阈值、聚类、马尔科夫随机场、区域增长、分裂合并、三维边缘检测、形态学、形变模型、水平集等方法。目前并没有单一的方法能解决图像分割的所有场景,全自动的分割方法通常针对特定器官和场景,当先验知识缺少时,全自动方法的效果会显著下降。因此,半自动的方法被广泛使用,该方法需要用户提供初值如轮廓、初始点信息等,并交互结果。图8给出了常用医学图像处理软件Slicer对胸部CT图像进行半自动分割的标注结果。GPU可以加速整个图像分割和可视化过程,从而提高图像分割的交互体验,进而实现更好的图像分割效果。
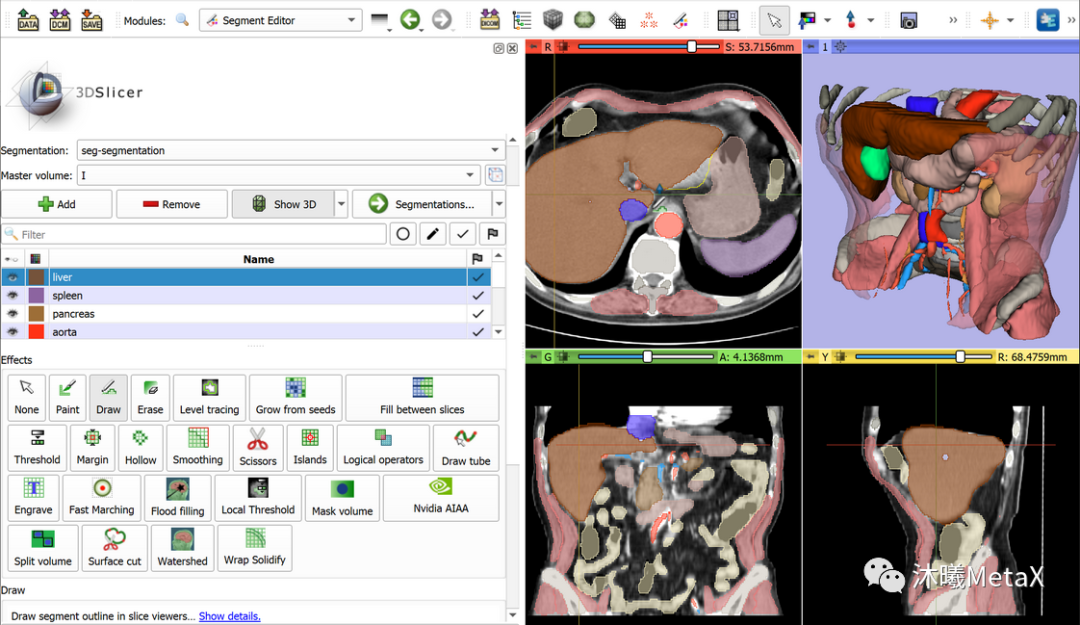
图8:利用软件Slicer对图像进行半自动分割标注
运动目标检测与跟踪
运动目标检测与跟踪通常分为两个阶段,首先是利用运动目标检测算法从视频或图像序列中把场景中的运动目标从背景中区分并提取出来,然后应用目标跟踪算法对该目标的运动特性及目标外观特征等参数进行分析和预测。除广泛用于智能视频监控领域之外,运动目标检测与跟踪在医学图像领域主要用于内窥镜视频中对微创手术 (Minimally Invasive Surgery) 器械进行检测与跟踪[7, 8]、时空细胞显微镜成像 (Spatial-Temporal Cell microscopy Imaging) 中对细胞运动、增殖的跟踪分析[9]、心脏磁共振成像 (Cine MRI) 或心动超声 (echocardiography) 中对心脏运动跟踪以检测与量化心脏功能[10]以及放射治疗4D-CT[11]中对肿瘤呼吸运动的跟踪等。运动目标检测与跟踪涉及大量2D或3D图像序列的分析计算,极为耗时,利用GPU并行加速可以实现较大加速比。图9展示了时空细胞显微镜成像分析中不同跟踪算法的单帧图像计算时长对比,可以看出随着计算量的增加,经过优化的GPU并行算法可以实现近166倍的加速比[9]。
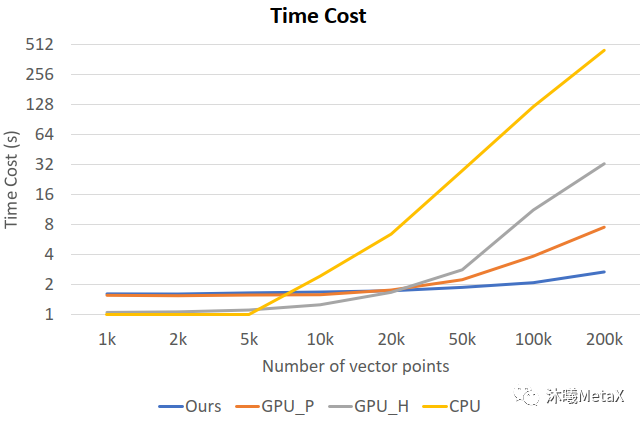
图9:不同跟踪算法的单帧图像计算时长。
横坐标为帧间图像的向量点数目,
纵坐标为单帧图像耗时;
GPU_H、CPU为传统跟踪算法的GPU、CPU版本,
GPU_P为并行张量加速算法,
Ours为文献[9]中提到的经优化的GPU加速算法。
来源:M. Zhao, A. Jha, Q. Liu, B. A. Millis, A. Mahadevan-Jansen, L. Lu, et al. Faster Mean-shift: GPU-accelerated clustering for cosine embedding-based cell segmentation and tracking. 2021
图像可视化
医学图像可视化是指利用医学影像设备获取的序列图像信息,重建三维图像模型,并将其转换成图形图像在屏幕上显示,为用户提供具有真实感并进行相互交互的三维医学图像。医学图像可视化广泛应用于临床诊断、放射治疗规划、手术导航、医学模拟仿真教学等方面,对临床医学具有很高的应用价值与实践意义。
根据绘制过程中数据描述方法的不同,医学图像可视化通常分为两大类——面绘制和体绘制。面绘制是指对体表面重建,然后用传统图形学技术实现表面绘制。面绘制可以有效地绘制出物体表面,但缺乏对物体内部信息的表达。由于面绘制技术的缺点和局限,体绘制(体渲染)技术应用更为广泛,其基本原理是利用光学模型模拟光线穿越半透明物质时的能量累积变化。具体而言,首先对每个体素赋以不透明度 (a) 和颜色值 (R、G、B) ;再根据各个体素所在点的梯度以及光照模型计算相应体素的光照强度;然后根据光照模型,将射线上投射到图像平面中同一个像素点的各个体素的不透明度和颜色值组合在一起,生成最终结果图像。
图像可视化计算量极大,对时间较为敏感,利用GPU的并行加速和纹理内存可以提高渲染速度。近年来随着虚拟现实 (Virtual Reality,VR) 技术在医学图像领域的兴起,图像可视化要求至少60帧每秒的速度和1024的高分辨率,这对GPU的内存和计算速度都提出了更高的要求。图10展示了利用VR对三维医学图像进行操作观察的示例[12]。
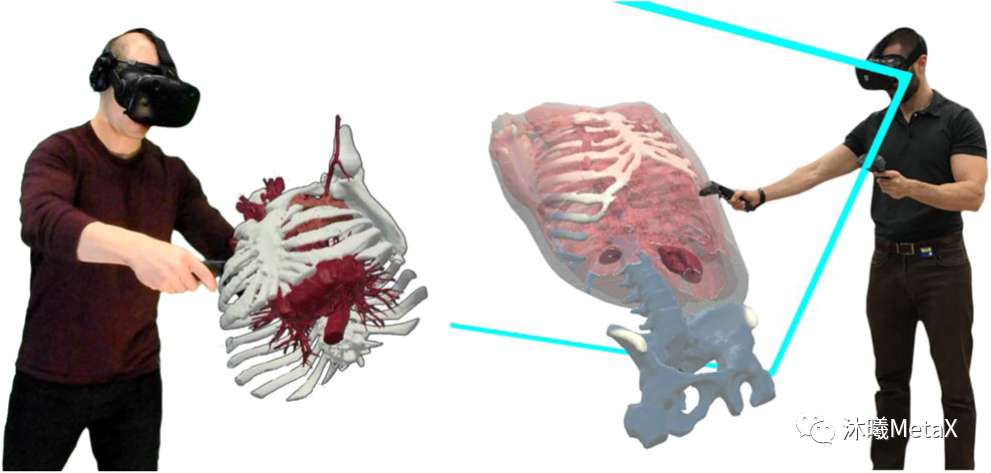
图10:医学图像VR操作示例
来源:J. Sutherland, J. Belec, A. Sheikh, L. Chepelev, W. Althobaity, B. J. Chow, et al. Applying modern virtual and augmented reality technologies to medical images and models. Journal of digital imaging, 2019
其他技术
医学图像处理还包括图像分类与识别、目标定位与检测等高层图像处理技术。其中,图像分类与识别,用于辅助诊断图像是否有病灶,并对病灶进行量化分级;图像目标定位用于识别图像特定目标,并给出具体物理位置;图像目标检测则需要把图像中所有目标识别出来,并确定其物理位置和类别。
这些处理技术传统上通常需要经过图像预处理、图像分割、特征提取、特征选择、分类器训练等过程。传统特征提取技术包括纹理、边缘等全局特征和SIFT (Scale-Invariant Feature Transfor,尺度不变特征变换) 、SURF (Speeded Up Robust Features,加速鲁棒性特征) 等局部特征。由于医学图像的多样性和复杂性,根据先验知识人工设计的特征往往只适合特定场景任务。传统图像分类、检测等方法不仅耗时耗力,需要大量的参数调节 (parameter hypertuning) 实验,计算量大,借助GPU加速计算,可以加快特征提取计算和相关算法的调参训练过程。
GPU赋能临床智能医疗
上文中,医学图像处理的各个任务流程都可以视作映射函数F的输入输出,即y=F(x) 。其中x为单个或多个图像(数字信号),y为输出,其定义取决于任务流程类型。例如,在图像重建中,y为高分辨率重建图像;在滤波增强中,y为图像质量增强图像;在图像配准中,y为配准参数;在图像分割中,y为图像蒙版mask。
在众多映射函数F的估计方法中,深度学习 (Deep Learning,DL)方法利用深度神经网络 (Deep Neural Network,DNN) 的多层非线性变换能实现映射函数F的无限逼近、表征输入数据分布,具有强大的自动特征提取和复杂模型构建能力,在医学图像处理领域应用十分广泛。其中,GPU是深度学习模型训练的算力保障。而一旦模型训练完成,借助GPU的高效前向运算,在图像重建、图像识别与分类、图像定位与检测、图像分割、图像配准和融合等方面,深度学习算法相比传统算法可以更为准确、高效地完成图像处理任务,辅助医生诊断治疗和预后。图11总结了深度学习在医学图像处理领域不同层次的分类。
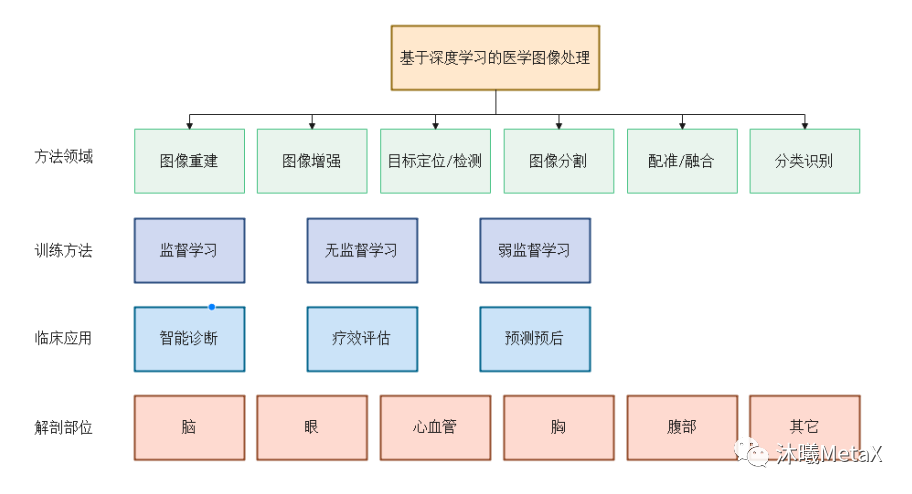
图11:基于深度学习的医学图像处理分类
深度学习在医学图像领域发展十分迅速,产生了许多性能优异的临床应用。例如,研究人员利用10万多幅恶性黑色素瘤(最致命的皮肤癌)和良性痣的图像训练了卷积神经网络(CNN)来识别皮肤癌,表现优于有经验的皮肤科医生,其漏诊率和误诊率都更低[13]。国内联影企业利用bi-c-GAN模型,从超低剂量的PET/MRI数据重建出高分辨率PET图像[14]。2020年COVID-19爆发早期,由于检测试剂的短缺,欧洲研究人员开发了CORADS-AI系统[15],利用CT图像筛查和评价COVID-19的感染情况,可以近实时(<2 s)得出结果。如图12所示,第一行为胸部CT图,第二行为利用CNN模型分割的肺结节解剖图,第三行为利用3D U-Net模型分割的具有毛状玻璃样病变的肺部区域,最下图为病变区域在正常结节部位的计算比例,左下图为基于感染比例得出的COVID-19感染分级和概率。
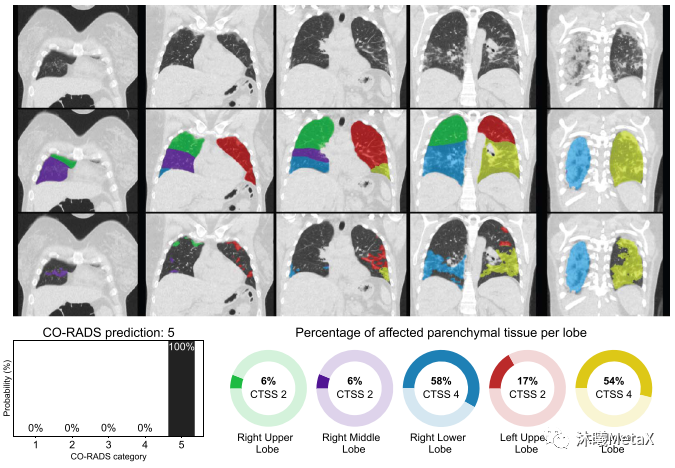
图12:CORADS-AI系统筛查COVID-19的结果示意图
来源:S. K. Zhou, H. Greenspan, C. Davatzikos, J. S. Duncan, B. Van Ginneken, A. Madabhushi, et al. A review of deep learning in medical imaging: Imaging traits, technology trends, case studies with progress highlights, and future promises. Proceedings of the IEEE, 2021
虽然深度学习在医学图像处理领域取得了高速的发展,但仍存在很多挑战和限制。除注册审批、市场准入等政策法规方面的挑战外,最大的挑战来自影像数据质量问题。医学影像数据的数量和质量决定了Al模型学习的结果。尽管医学图像数据总量巨大,但针对不同病种的影像数据量和质量参差不齐,有些病种的训练数据缺乏;健康大数据孤岛问题(即医院间医疗数据不能共享和联通问题)、安全隐私问题仍然存在,虽然有所缓解,但仍未达到深度学习的要求。医疗影像数据针对细分场景的数据量和质量仍无法满足算法模型的训练需求。影像数据处理结果需要大量影像科专业医师去标注,成本、耗时巨大,而医师的经验水平、学习数据的质量和典型性都将影响数据的标注质量。针对这一问题,需要建立完善的医学数据共享和隐私学习平台,利用大数据技术从海量数据中筛选收集研究所需影像数据,并借助自动化技术辅助医师标注,其涉及的大量运算需要GPU进行算力支持。而针对小样本数据集,可以通过数据增强、迁移学习 (Transfer Learning) 、生成对抗网络 (Generative Adversarial Network,GAN) 、域适应 (Domain Adaptation) 、自监督学习 (Self-supervised Learning) 、弱监督学习 (Weakly Supervised Learning) 等方法在一定程度上缓解训练样本不足的问题。
目前深度学习算法普遍存在鲁棒性和泛化性不足的问题,其研发的模型很有可能仅对训练数据表现良好。而由于成像角度、成像噪声、重建算法等因素的影响造成测试数据集和训练数据集具有不同的特点,训练模型对这类数据预测完全失效。针对该问题,除扩大训练数据的代表性外,还可以探索模型结构,提高模型深度,引入对抗学习、注意力、多任务等机制,以提升模型泛化能力。这些复杂模型的训练和预测都依赖更高的GPU算力和显存。
此外,深度学习模型是一个黑盒子,可解释性较差。即使经过训练的模型在测试数据集显示出很高的准确性,临床医生仍会对定期使用它们分析患者数据持谨慎怀疑态度。因此,模型的解释和不确定性量化研究十分必要。深度学习模型的可解释性主要包括模型结构的可解释性、特征权重的统计分析以及利用可解释模型如决策树近似模拟深度学习模型等[16]。而模型的不确定性量化,则是将多种不确定性来源和个体差异性融入模型预测中,给出模型预测准确程度的置信区间。模型的解释和不确定性量化,有助于增强深度学习结果的可信度,从而加速人工智能在医学图像临床应用的落地。
总结
医学图像在疾病诊断、手术治疗、预后评估等临床应用中起着举足轻重的作用,超过70%的诊断都依赖医学图像。对医学图像的处理分析,有助于促进自动化诊疗过程,是推动精准医疗和个性化医疗的重要工具。由于医学图像处理任务的复杂性和多样性,以及图像分辨率的提高,医学图像处理的算法复杂且运算量巨大,利用GPU可以实现高性能并行计算。
近年来,人工智能逐渐成为医学影像发展的主流方向,相比传统方法,可以提供更为准确、高效的处理结果。但由于图像数据质量的问题以及模型泛化性的要求,深度学习模型的结构和训练也愈发复杂,这对GPU的算力和显存都提出了更高的要求。
我们预计,随着成像设备的升级、医学图像标记数据的不断累积以及深度学习算法的不断完善,人工智能和GPU的结合将在医学图像处理领域展现更为广阔的未来。
[1] J. Wu. The Next Era: Flourish of National Health Care & Medicine with the World Leading Artificial Intelligence. Chinese Medical Sciences Journal, 2019, 34(2): 69-70
[2] S. Nam, M. Akçakaya, T. Basha, C. Stehning, W. J. Manning, V. Tarokh, et al. Compressed sensing reconstruction for whole‐heart imaging with 3D radial trajectories: a graphics processing unit implementation. Magnetic resonance in medicine, 2013, 69(1): 91-102
[3] S. Cuomo, P. De Michele, F. Piccialli. 3D Data Denoising via Nonlocal Means Filter by Using Parallel GPU Strategies. Computational and Mathematical Methods in Medicine, 2014, 2014: 523862
[4] S. Ekström, M. Pilia, J. Kullberg, H. Ahlström, R. Strand, F. J. J. o. M. I. Malmberg. Faster dense deformable image registration by utilizing both CPU and GPU. 2021, 8(1): 014002
[5] S. A. Mahmoudi, M. A. Belarbi, E. W. Dadi, S. Mahmoudi, M. J. C. Benjelloun. Cloud-based image retrieval using GPU platforms. 2019, 8(2): 48
[6] 曹强. 多模医学图像融合算法及其在放疗中的应用研究[博士]. 东南大学, 2021
[7] Y. Jin, Y. Yu, C. Chen, Z. Zhao, P.-A. Heng, D. J. I. T. o. M. I. Stoyanov. Exploring Intra-and Inter-Video Relation for Surgical Semantic Scene Segmentation. 2022,
[8] D. Bouget, M. Allan, D. Stoyanov, P. Jannin. Vision-based and marker-less surgical tool detection and tracking: a review of the literature. Medical image analysis, 2017, 35: 633-654
[9] M. Zhao, A. Jha, Q. Liu, B. A. Millis, A. Mahadevan-Jansen, L. Lu, et al. Faster Mean-shift: GPU-accelerated clustering for cosine embedding-based cell segmentation and tracking. 2021, 71: 102048
[10] E. Ferdian, A. Suinesiaputra, K. Fung, N. Aung, E. Lukaschuk, A. Barutcu, et al. Fully Automated Myocardial Strain Estimation from Cardiovascular MRI–tagged Images Using a Deep Learning Framework in the UK Biobank. Radiology: Cardiothoracic Imaging, 2020, 2(1): e190032
[11] P. Jafari, S. Dempsey, D. A. Hoover, E. Karami, S. Gaede, A. Sadeghi-Naini, et al. In-vivo lung biomechanical modeling for effective tumor motion tracking in external beam radiation therapy. Computers in Biology and Medicine, 2021, 130: 104231
[12] J. Sutherland, J. Belec, A. Sheikh, L. Chepelev, W. Althobaity, B. J. Chow, et al. Applying modern virtual and augmented reality technologies to medical images and models. Journal of digital imaging, 2019, 32(1): 38-53
[13] H. A. Haenssle, C. Fink, R. Schneiderbauer, F. Toberer, T. Buhl, A. Blum, et al. Man against machine: diagnostic performance of a deep learning convolutional neural network for dermoscopic melanoma recognition in comparison to 58 dermatologists. Annals of oncology, 2018, 29(8): 1836-1842
[14] H. Sun, Y. Jiang, J. Yuan, H. Wang, D. Liang, W. Fan, et al. High-quality PET image synthesis from ultra-low-dose PET/MRI using bi-task deep learning. Quantitative Imaging in Medicine Surgery, 2022, 12(12): 5326-5342
[15] M. Prokop, W. Van Everdingen, T. van Rees Vellinga, H. Quarles van Ufford, L. Stöger, L. Beenen, et al. CO-RADS: a categorical CT assessment scheme for patients suspected of having COVID-19—definition and evaluation. Radiology, 2020, 296(2): E97-E104
[16] 化盈盈, 张岱墀, 葛仕明. 深度学习模型可解释性的研究进展. Journal of Cyber Security 信息安全学报, 2020, 5(3)
编辑:黄飞
 电子发烧友App
电子发烧友App
























评论